Table des matières:
- Étape 1: Organisation des données de mammographie
- Étape 2: Traitement d'image
- Étape 3: Code de seuil
- Étape 4: Recherche d'anomalies pour chaque image binaire
- Étape 5: Tracer l'emplacement et la taille de la masse diagnostiquée pour une comparaison visuelle
- Étape 6: mise en œuvre de la deuxième méthode de comparaison
- Étape 7: Analyser les données collectées
- Étape 8: Créer votre propre classificateur
- Étape 9: Des améliorations ? Des pensées?
- Auteur John Day day@howwhatproduce.com.
- Public 2024-01-30 09:08.
- Dernière modifié 2025-01-23 14:46.
Le but de ce projet était d'identifier et d'utiliser un paramètre pour traiter des images de mammographie en niveaux de gris de diverses classifications de tissus de fond: gras, gras glandulaire et tissu dense. Cette classification est utilisée lorsque les radiologues analysent des mammographies et doivent déterminer si la densité tissulaire masquera des anomalies telles que des lésions ou des tumeurs. C'est parce que les deux structures physiologiques normales telles que le tissu glandulaire et le tissu conjonctif fibreux. et les morphologies anormales telles que les calcifications et les tumeurs apparaîtront très lumineuses sur la mammographie tandis que les tissus adipeux moins denses apparaîtront en noir. Par conséquent, il était approprié de programmer un classificateur capable de manipuler les niveaux d'intensité des pixels pour mieux visualiser et identifier les masses.
Étape 1: Organisation des données de mammographie
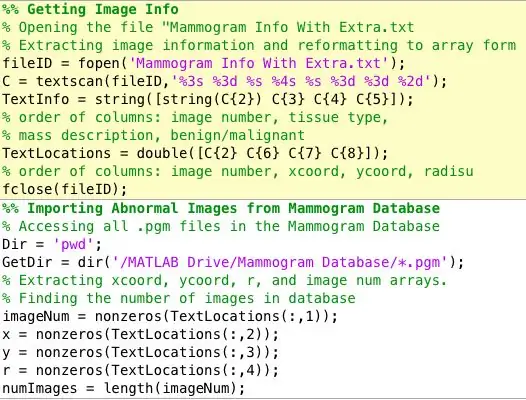
L'une des premières choses que j'ai réalisé que je devais gérer était d'organiser les données d'une manière très claire, concise et accessible. Ce sont les variables que j'ai extraites de la base de données mini-MIAS des mammographies. J'ai créé deux tableaux. Un contenant 4 colonnes:
- Numéro d'image:
- x coordonnée de masse
- y coordonnée de masse
- Rayon de masse: (Ceci a défini une taille approximative pour la masse
Le deuxième tableau contenait des informations de classification:
- Type de tissu de fond: gras (F), gras glandulaire (G), dense (D)
- Description de la masse: bien définie (CIRC), spiculée (SPIC), autre mal définie (MISC) Distorsion architecturale (ARCH), asymétrie (ASYM), normale (NORM)
- Diagnostics: bénin (B), malin (M)
Étant donné que le but de ce projet était de déterminer le meilleur seuil pour chaque type de tissu de fond, toutes les informations n'étaient pas nécessaires. Cependant, vous pouvez étendre votre projet pour inclure l'analyse de texture et tester votre classificateur par rapport aux descriptions de masse connues.
Note latérale: La base de données à partir de laquelle j'ai obtenu les images de mammographie diagnostiquées a organisé les informations sur chaque mammographie dans un fichier texte séparé des images. Il m'a été légèrement difficile d'extraire les données d'un fichier texte et de les organiser sous forme de tableau, mais le lien suivant a été très utile pour comprendre tout cela. Alternativement, ajustez simplement le code que j'ai collé ci-dessus pour vos besoins.
Format de fichier de mammographie: mdb001 G CIRC B 535 425 197
mdb002 G CIRC B 522 280 69
Aide TextScan: https://www.mathworks.com/help/matlab/ref/textsca…Base de données sur les mammographies:
Étape 2: Traitement d'image
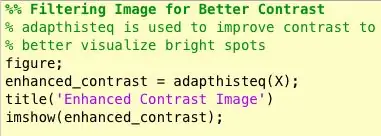
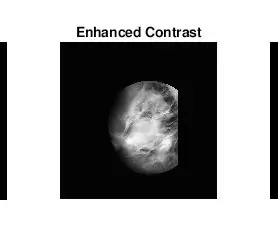
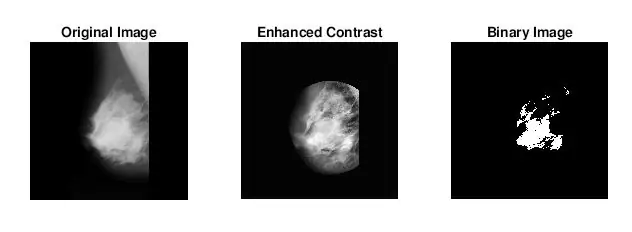
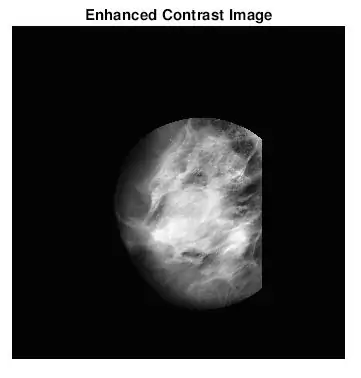
Eh bien, la deuxième chose qui m'est venue lorsque je cherchais à identifier les masses était que pour de nombreuses mammographies anormales, je ne pouvais pas dire visuellement où se trouvait l'anomalie ou quelle était sa taille. Évidemment, comme je ne suis pas un radiologue expérimenté, c'était prévu. Cependant, le moyen le plus simple de trouver des anomalies (selon mes longues recherches sur Google) consistait à examiner les concentrations de zones claires et sombres. J'ai principalement utilisé la fonction adapthisteq pour améliorer le contraste de l'image, puis imbinarize pour convertir l'image en une image binaire afin d'expérimenter différents niveaux de seuil.
- adapthisteq: Cette fonction transforme les valeurs d'intensité des images en niveaux de gris et rvb à l'aide d'une égalisation d'histogramme adaptative à contraste limité. En d'autres termes, il ajuste l'histogramme des valeurs d'intensité à un type de distribution spécifié. Le lien mathworks pour cette fonction est joint ci-dessous pour une lecture plus approfondie.
- imbinarize: crée une image binaire à partir d'une image en niveaux de gris en attribuant tous les pixels au-dessus d'une certaine intensité à 1s et les pixels en dessous de cette valeur à 0. J'ai utilisé cette fonction pour tester le seuil optimal pour réduire le bruit de fond des tissus.
Étape 3: Code de seuil
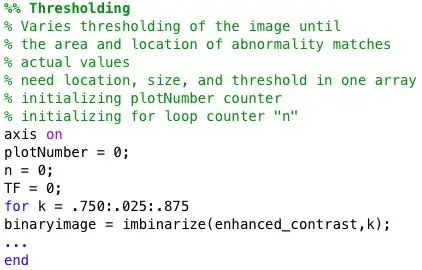
Une boucle for est utilisée pour binariser la mammographie avec des seuils variables. Pour donner une vue d'ensemble, la boucle for contient le code de l'étape 3 à l'étape 7. Ainsi, chaque image binaire sera analysée à la recherche d'anomalies. De plus, cette boucle for est enfermée dans une autre boucle for qui importe une nouvelle image de mammographie de la base de données à chaque itération.
Étape 4: Recherche d'anomalies pour chaque image binaire
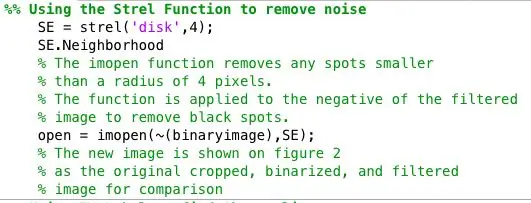
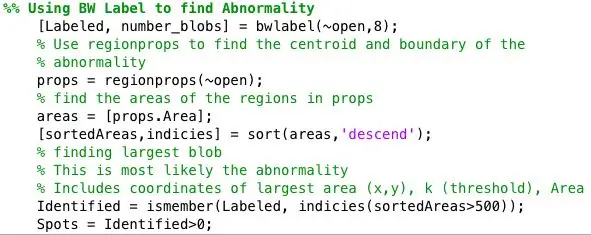
J'ai ensuite traité les images binaires en utilisant la fonction strel en conjonction avec imopen pour supprimer le bruit de fond. L'image binaire de l'étape précédente est inversée et filtrée en utilisant le voisinage défini par SE. Ensuite, j'ai utilisé bwlabel pour étiqueter toutes les zones qui avaient au moins 8 pixels connectés.
La fonction region props a été utilisée pour trouver les propriétés centroïdes et de surface de chaque tache identifiée par bwlabel.
Ensuite, tous les points de plus de 500 pixels ont été identifiés à l'aide d'ismember. Les centroïdes des taches identifiées ont été tracés sur une image qui n'affichait que les taches d'une superficie supérieure à 500. Area Identified = ismember(Labeled, indicies(sortedAreas>500)); Taches = Identifiées>0;
Étape 5: Tracer l'emplacement et la taille de la masse diagnostiquée pour une comparaison visuelle
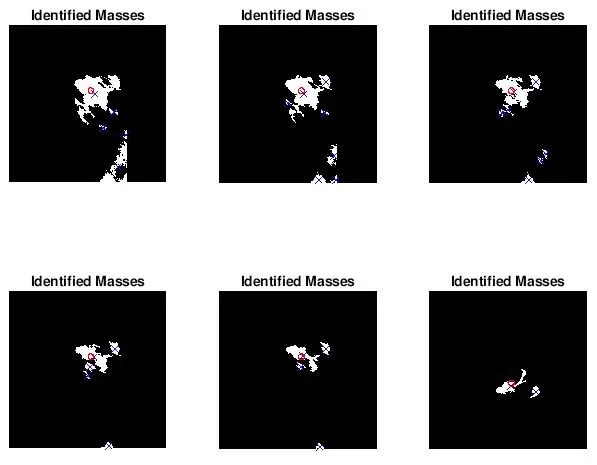
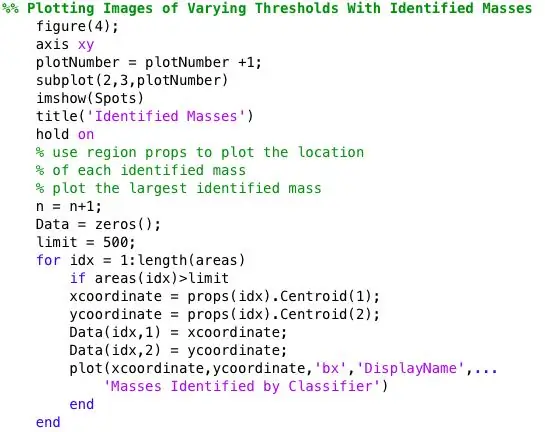

Je voulais voir si les spots trouvés par bwlabel étaient corrects. Je l'ai fait de deux manières. J'ai d'abord analysé la précision de mon classificateur en faisant une comparaison visuelle. J'ai simplement tracé la taille et l'emplacement réels de l'anomalie (cercle rouge) et l'emplacement déterminé par le code (x bleu) sur l'image de mammographie prétraitée. Les six images ci-dessus montrent les effets de l'augmentation de la valeur seuil en niveaux de gris.
Étape 6: mise en œuvre de la deuxième méthode de comparaison

La deuxième façon dont j'ai testé le classificateur et les valeurs de seuil consistait à déterminer si les emplacements trouvés par le classificateur se trouvaient à une certaine distance des coordonnées de l'anomalie diagnostiquée. J'ai enregistré les seuils pour lesquels au moins un des points identifiés était à moins de 1,5 * r de l'anomalie connue dans un fichier texte séparé appelé Mammogram Data. Le but était de trouver le seuil minimum nécessaire à mon classificateur pour identifier l'anomalie.
Étape 7: Analyser les données collectées
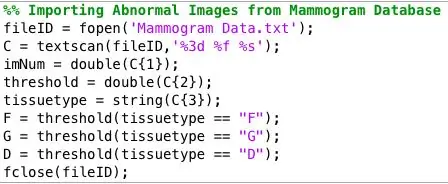
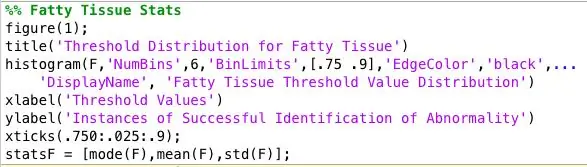
J'ai exécuté le programme sur toutes les images de mammographie anormales et je me suis retrouvé avec un énorme fichier texte de données. Afin de trouver le meilleur seuil pour chaque type de tissu, j'ai organisé les données par type de tissu et tracé un histogramme des valeurs de seuil pour chaque type de tissu. La valeur seuil appropriée a été décidée en fonction du seuil qui fournissait les résultats les plus précis pour chaque type de tissu. J'ai enregistré ces données pour les télécharger dans mon classificateur.
Étape 8: Créer votre propre classificateur

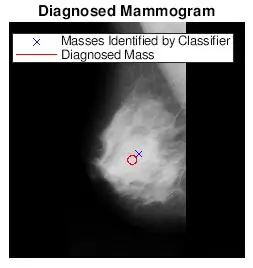
Après avoir trouvé les valeurs de seuil les plus appropriées pour chaque type de tissu, j'ai modifié mon code d'origine pour qu'un utilisateur saisisse le numéro d'image et le type de tissu afin de choisir le seuil pour l'image de mammographie. J'ai ensuite tracé l'emplacement de la mammographie diagnostiquée avec les emplacements trouvés sur les images de mammographie originales. Je voulais rendre cela plus amusant alors j'ai programmé une fonction pour recadrer une région circulaire entourant le retour sur investissement. L'utilisateur serait invité à choisir un point central et plusieurs points qui englobent le mieux le retour sur investissement. J'ai joint les deux fichiers matlab ici.
Étape 9: Des améliorations ? Des pensées?
Au moment où j'écrivais ce instructable, je commence à voir de nombreuses améliorations que je pourrais apporter au classificateur, telles que trouver des moyens de distinguer les différents types de masses identifiés en fonction de l'analyse de la texture ou améliorer mes tests de précision dans la section SandBoxProject. déposer. Comme il s'agissait d'un projet avec une date limite, j'ai dû m'arrêter quelque part, mais j'espère pouvoir utiliser les compétences en traitement d'images que j'ai acquises dans d'autres applications. De plus, j'ai joint le fichier qui a été utilisé pour traiter par lots toutes les images de mammographie anormales.
Conseillé:
Visualiser les données de Magicbit dans AWS : 5 étapes

Visualisation des données de Magicbit dans AWS : les données collectées à partir des capteurs connectés à Magicbit seront publiées sur le noyau AWS IOT via MQTT pour être visualisées graphiquement en temps réel. Nous utilisons magicbit comme carte de développement dans ce projet basé sur ESP32. Donc tout ESP32 d
Utilisation de la carte Complex Arts Sensor pour contrôler les données pures via le WiFi : 4 étapes (avec images)

Utilisation de la carte Complex Arts Sensor pour contrôler les données pures via le Wi-Fi : avez-vous déjà voulu expérimenter le contrôle gestuel ? Faire bouger les choses d'un geste de la main ? Contrôler la musique d'un simple mouvement du poignet ? Ce Instructable va vous montrer comment ! Le Complex Arts Sensor Board (complexarts.net) est un microc
Gérez 100 appareils dans les applications, les organisations, les rôles et les utilisateurs Ubidots : 11 étapes

Gérer 100 appareils dans les applications, les organisations, les rôles et les utilisateurs d'Ubidots : à cette occasion, j'ai décidé de tester la fonctionnalité de la plate-forme IoT Ubidots pour la gestion des utilisateurs dans le cas où elle aurait de nombreux appareils pour plusieurs organisations ou entreprises sous la même Plateforme Ubidots. Information du maire : vous
N : Comment faire une sculpture acrylique et LED multicouche avec des niveaux d'éclairage variables : 11 étapes (avec photos)

N : Comment faire une sculpture multicouche en acrylique et LED avec des niveaux d'éclairage variables : Ici, vous pouvez découvrir comment vous faire votre propre n comme réalisé pour l'exposition www.laplandscape.co.uk organisée par le groupe d'art/design Laponie. Plus d'images peuvent être vues sur flickrCette exposition se déroule du mercredi 26 novembre au vendredi 12 décembre 2008 inclusi
Écoutez vos morceaux pour travailler dans des niveaux de bruit élevés. : 16 étapes

Écoutez vos airs pour travailler dans des niveaux de bruit élevés. : C'est ma première fissure à ce sujet si nu avec moi. Problème : au travail, nous n'avons pas le droit de porter des écouteurs et vu qu'il existe une règle de protection auditive à 100%, c'est ainsi que je bats le système. Nous avons le droit aux radios mais nous sommes dans un bâtiment en acier et ra
