Table des matières:
- Étape 1: Utilisation de Brain Box
- Étape 2: Introduction: Analyse des coupes transversales
- Étape 3: Configuration d'une fonction: LevelCurveTracings.m
- Étape 4: Développement de l'algorithme lié: Recherche de limites
- Étape 5: Développement de l'algorithme lié: génération d'un tableau externe lié
- Étape 6: Développement de l'algorithme lié: Utilisation du point central
- Étape 7: Développement de l'algorithme lié: lorsqu'un centroïde n'est pas centré
- Étape 8: Développement de l'algorithme lié: interférence des trous
- Étape 9: Développement de l'algorithme lié: localisation des trous, finalisation des limites du cerveau et des limites des trous
- Étape 10: Enregistrement des données: Fonction PatientFiles.m
- Étape 11: enregistrement des données dans des fichiers
- Étape 12: Enregistrement des données: affichage d'un graphique du volume cérébral au fil du temps
- Étape 13: Combler les lacunes dans les sous-parcelles: Subplotclose.m
- Étape 14: Le code principal: Tout effacer et demander des entrées
- Étape 15: Le code principal: traitement par lots des images
- Étape 16: Le code principal: le rembourrage
- Étape 17: Le code principal: Détermination des limites
- Étape 18: Le code principal: déterminer la bonne dimension Z
- Étape 19: Le code principal: Détermination des coordonnées X et Y
- Étape 20: Le code principal: tracer une structure tridimensionnelle, trouver un volume et enregistrer des données
- Auteur John Day day@howwhatproduce.com.
- Public 2024-01-31 10:19.
- Dernière modifié 2025-01-23 14:46.
L'avancée vers la frontière d'une vie humaine plus longue a provoqué la montée de maladies que les civilisations avant la nôtre n'avaient pas vues. Parmi ceux-ci, la maladie d'Alzheimer a touché environ 5,3 millions d'Américains âgés vivants en 2017, soit environ 1 Américain âgé sur 10 (https://www.alz.org/facts/) et d'innombrables autres personnes atteintes de démence. Pour aider à lutter pour comprendre ce qui afflige nos aînés, ce code dotera les futurs chercheurs et curieux curieux de la capacité de suivre le volume cérébral au fil du temps.
Étape 1: Utilisation de Brain Box
Pour utiliser brain box, il suffit de:
- IRM d'un cerveau et le nom et le format de ces fichiers (devraient tous avoir à peu près les mêmes dimensions)
- Longueur d'un scan
- Distance entre chaque couche (scan IRM)
- Nom du patient (Lors de la saisie, n'incluez pas d'espaces et veuillez mettre le prénom et le nom en majuscule, comme par exemple: PrénomNom)
Et à partir de là, on a la capacité de suivre les tendances du volume cérébral d'un individu au fil du temps. Ainsi, les chiffres des tendances d'Alzheimer peuvent être suivis par ce logiciel. La longueur que nous avons utilisée dans l'essai était de 180 mm pour la longueur d'un scan et de 5 mm pour la distance entre les scans IRM, sur la base de chiffres moyens.
Cependant, l'application de brain box n'a pas besoin d'être limitée à cette seule tâche. Si les sections transversales d'un solide donné sont photographiées, comme une tumeur en soi, les tendances des changements de volume pour ceux-ci peuvent également être suivies dans le logiciel.
Étape 2: Introduction: Analyse des coupes transversales
Dans les structures tridimensionnelles, les plans bidimensionnels qui en sont constitués seraient appelés sections transversales. Imaginez qu'une pile de papiers forme un prisme rectangulaire, alors chaque morceau de papier serait une coupe transversale du papier. En imaginant le cerveau, nous appliquons le même cours de pensée. L'IRM (imagerie par résonance magnétique) (voir les informations sur l'IRM) capture les coupes transversales du cerveau, et en utilisant les limites définies dans chaque "couche" du cerveau fournie, nous pouvons construire une structure pour modéliser et trouver le volume du cerveau. Cependant, nous devons d'abord construire une fonction pour fournir des informations sur ces limites.
Étape 3: Configuration d'une fonction: LevelCurveTracings.m
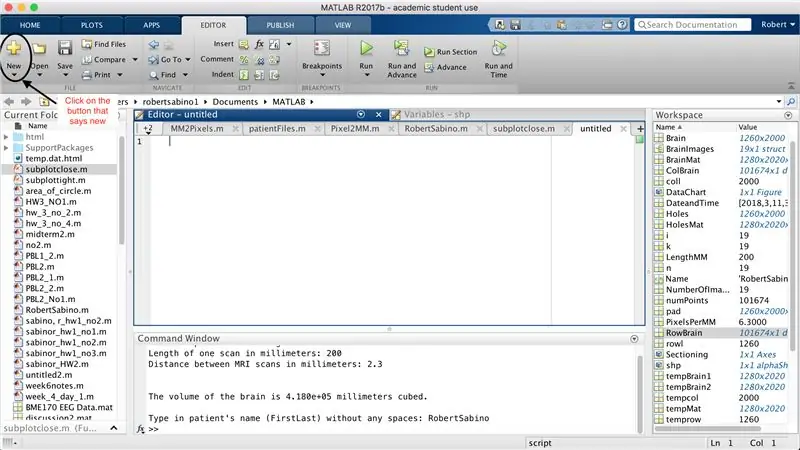
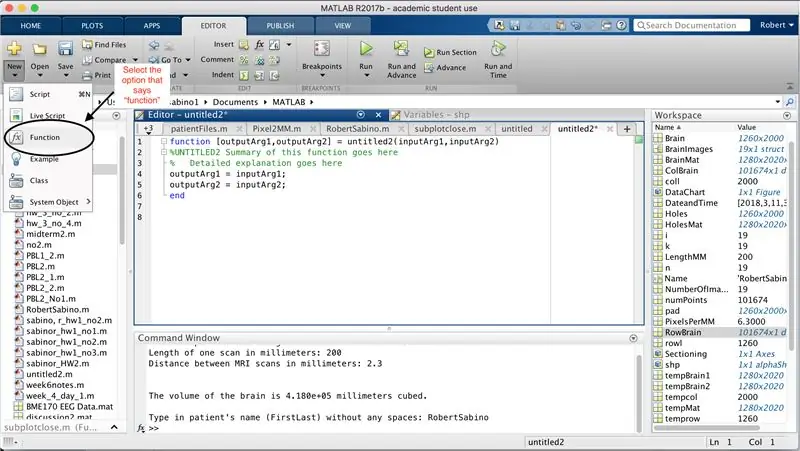
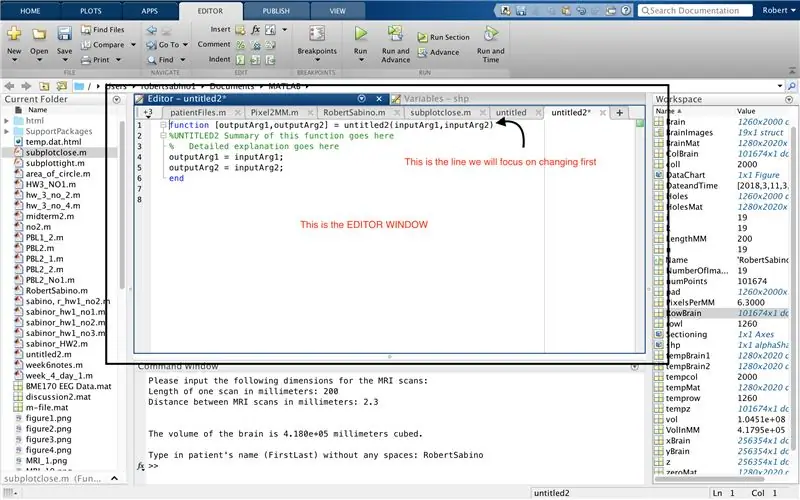

Tout d'abord, assurez-vous que votre ordinateur a téléchargé MATLAB_R2017b (télécharger ici) et ouvrez MATLAB. Dans l'interface MATLAB, cliquez sur le bouton dans le coin supérieur gauche de la fenêtre qui dit "Nouveau" avec un signe plus jaune gras, et sélectionnez l'option "fonction", pour ouvrir dans la fenêtre de l'éditeur un espace qui ressemble à celui du troisième photo. Nous allons nous concentrer sur la modification de la première ligne pour configurer la fonction. Où il est écrit "outputArg1", remplacez-le par "brain", "outputArg2" pour dire "holes", "untitled2" à "exp2" et "inputArg1" à "image", et supprimez "inputArg2". Vous avez maintenant une fonction à appeler en utilisant "exp2", en prenant un argument "image" et en affichant les limites de "cerveau" et "trous". La première ligne de la fonction doit ressembler à la ligne représentée dans la quatrième image. Supprimez tout le code sous cette première ligne.
Étape 4: Développement de l'algorithme lié: Recherche de limites

Tapez le code comme suit sous la ligne. Cette section de la fonction effectue les opérations suivantes ligne par ligne.
- Chargez l'image "image" dans la variable "mri".
- Transformez « irm » en une image composée de valeurs dans une plage de nombres à des uns et des zéros (alias binarisation) en fonction d'une valeur seuil définie. Si la valeur d'un pixel est égale ou supérieure à 0,1, elle est définie sur un, sinon, la valeur de ce pixel est définie sur zéro.
- Les quatre lignes suivantes transforment 10 colonnes et lignes sur les bords de la couche de balayage IRM en zéros, pour éviter de lire des valeurs incorrectes comme faisant des limites (comme appris en expérimentant avec le code).
- Dans la dernière ligne, bwboundaries trace les bornes de l'image binarisée "mri" et la met égale à "b", un tableau avec les éléments dont les indices correspondent à ceux des bornes fixées à un.
Étape 5: Développement de l'algorithme lié: génération d'un tableau externe lié
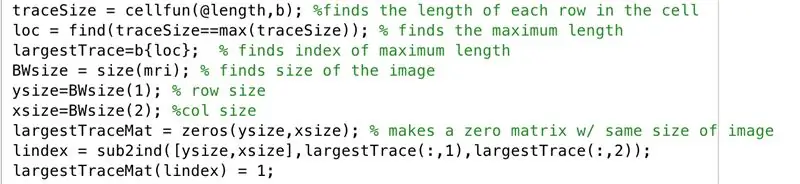
Suivez dans la fenêtre de l'éditeur avec le code suivant dans l'image. Cette section du code effectue les opérations suivantes ligne par ligne.
- Trouvez la longueur de chacune des lignes de l'image binarisée "b" (cellfun applique la longueur de la fonction à chaque ligne).
- Réglez "loc" pour stocker les longueurs maximales.
- Trouvez l'index de la longueur maximale, défini pour être stocké dans "largestTrace".
- Trouvez la taille de l'image "mri", qui est de la même taille que "b", et définissez-la sur "BWsize".
- Recherchez le nombre de lignes dans le tableau de l'image, défini sur "ysize".
- Trouvez le nombre de colonnes dans le tableau de l'image, défini sur "xsize".
- Générez le tableau "largestTraceMat", une matrice "ysize" par "xsize" de zéros.
- Trouvez l'indice équivalent à partir des valeurs indicées correspondant à l'endroit où se trouvaient les valeurs les plus grandes de Trace x et y, stockez-les dans le vecteur "lindex".
- Dans la matrice de zéros, "largestTraceMat", transformez les éléments aux indices qui correspondent aux valeurs d'indice stockées en tant qu'éléments dans "lindex" en uns.
Ainsi, le tableau logique "largestTraceMat" a la plus grande région délimitée de la section transversale donnée du scanner cérébral tracée comme un avec un fond de zéros
Étape 6: Développement de l'algorithme lié: Utilisation du point central

Ensuite, nous devons tester pour voir si la section transversale se compose de plus d'une région (la plus grande). En testant l'alignement du centroïde de la plus grande région, nous pouvons voir s'il existe une région contiguë, ce qui donnerait un centroïde plus centré, ou la possibilité de plusieurs régions.
- Utilisez "regionProps" pour trouver des informations sur les centroïdes présents, définis égaux au tableau de structure "tempStruct"
- Formez un tableau « centroïdes » avec les données du champ « centroïde » concaténées verticalement
- Prenez les valeurs de la deuxième colonne des "centroïdes" (les coordonnées de la dimension horizontale)
- Exécutez un filtre pour vérifier l'alignement du centre de gravité au centre horizontal
Étape 7: Développement de l'algorithme lié: lorsqu'un centroïde n'est pas centré
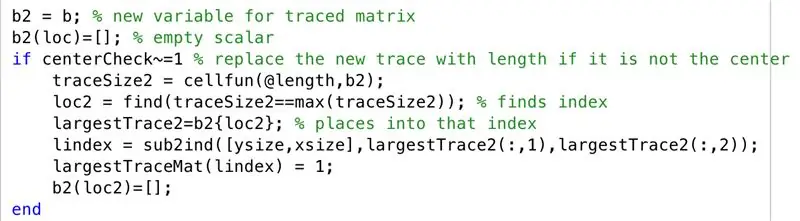
Dans le cas où le centroïde de la plus grande région de trace n'est pas centré, nous passons par les étapes suivantes. Comme nous l'avions observé dans les IRM, la tendance était d'avoir des hémisphères du cerveau représentés dans la section transversale lorsqu'ils ne sont pas contigus, nous continuons donc maintenant à tracer la deuxième plus grande trace avec la plus grande trace dans "largestTraceMat"
- Définissez la matrice tracée sur une nouvelle variable "b2"
- Initialiser la matrice vide "b2", avec un ensemble indexé par "loc"
- Créez un conditionnel, pour quand un centroïde n'est pas centré (c'est-à-dire une couche à plusieurs régions)
- Définir une nouvelle taille de trace à rechercher pour chaque ligne (traceSize2)
- Définir "loc2" pour trouver les indices où les limites sont présentes
- Laissez les cellules spécifiées par "loc2" dans "b2" être égales à "largestTrace2"
- Convertir les indices en indices, définir sur "lindex"
- Remplacez les éléments correspondant à "lindex" dans "largestTraceMat" par 1
- Initialiser la matrice vide "b2", avec un ensemble indexé par "loc2"
Étape 8: Développement de l'algorithme lié: interférence des trous

En traitant les trous, les valeurs stockées dans "b2" ont gardé une trace des structures autres que la plus grande trace, et les tracer sur une forme remplie de "largestTraceMat" révélera où il y a des trous dans les régions du cerveau.
- Créez un tableau "filledMat", qui est une forme remplie de "largestTraceMat"
- Créer un tableau "interferenceMat", un tableau de zéros "ysize" par "xsize"
- Créer un tableau "interferenceloc", pour stocker les valeurs de "b2", concaténées verticalement
- Créer un tableau "lindex" pour stocker les indices qui correspondent à "interferenceloc"
- Pour les indices dans "interferenceMat" qui correspondent à "lindex", définissez la valeur sur 1, créant une région délimitée différente
Étape 9: Développement de l'algorithme lié: localisation des trous, finalisation des limites du cerveau et des limites des trous

- Définissez le tableau "tempMat" égal à "interferenceMat" plus "filledMat", ajoutant ainsi chaque valeur de la matrice les unes aux autres
- Définir le tableau "holesLoc" égal aux indices où "interferenceMat" et "filledMat" étaient tous deux égaux à un
- Configurez "holesMat" comme une matrice zéro de dimensions "ysize" x "xsize"
- Définir des indices dans "holesMat" qui sont égaux à "holesLoc" comme un
- Définissez « cerveau » sur « largestTraceMat »
- Réglez "holes" sur "holesMat"
Avec la découverte où les valeurs des matrices ajoutées étaient égales à 2, les emplacements des trous ont été facilement sécurisés et tracés sur une matrice vide.
Étape 10: Enregistrement des données: Fonction PatientFiles.m
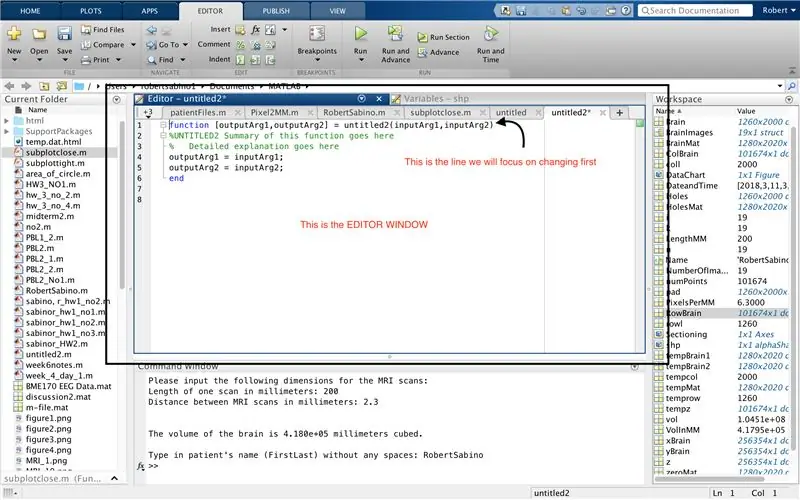

Tout comme la configuration de la dernière fonction, cliquez sur le bouton dans le coin supérieur gauche de la fenêtre qui dit "Nouveau" avec un signe plus jaune gras, et sélectionnez l'option "fonction", pour ouvrir dans la fenêtre de l'éditeur un espace qui ressemble à celui de la troisième photo. Dans la première ligne, supprimez la matrice de sortie et remplacez par simplement "output", remplacez "untitled2" par "patientFiles", supprimez tous les arguments d'entrée et suivez à la place le formatage spécifié dans la quatrième image de la ligne de code. La première ligne de cette fonction doit correspondre au formatage de l'image.
Étape 11: enregistrement des données dans des fichiers
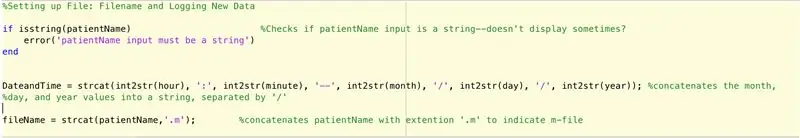

Pour mettre en place un fichier pour enregistrer les données trouvées par la fonction principale (encore à décrire), nous devons suivre ces étapes (comme prescrit par le code ligne par ligne).
- Vérifiez si l'entrée pour patientName est une chaîne.
- S'il ne s'agit pas d'une chaîne, affichez que l'entrée patientName doit être une chaîne.
- Terminer l'instruction if (éviter les erreurs).
- Configurez une instruction de chaîne "DateandTime" qui donnera le format suivant: heure:minutes--mois/jour/année.
- Définissez la variable fileName sur ce qui suit: patientName.m.
Passons maintenant à la section suivante de la fonction: un fichier de ce nom existe-t-il déjà ?
1) Supposons que le fichier de ce nom existe déjà:
- Exécutez le fichier pour obtenir les valeurs du passé en file d'attente
- Ajouter les données "DateandTime" de l'itération en cours en tant que nouvelle cellule dans le tableau de cellules de valeurs x (index end+1)
- Ajouter la valeur actuelle "brainVolume" en tant que nouvelle cellule dans le tableau de cellules de valeurs y (index end+1)
- Enregistrez les variables actuelles chargées dans le fichier.
2) Supposons que le fichier de ce nom n'existe pas:
- Créez un nouveau fichier avec le nom stocké dans la variable "patientName"
- Ajouter les données "DateandTime" actuelles en tant que cellule dans le tableau de cellules vides de valeurs x
- Ajouter les données "brainVolume" actuelles en tant que cellule dans le tableau de cellules vides de valeurs y
- Enregistrez les variables actuelles chargées dans le fichier.
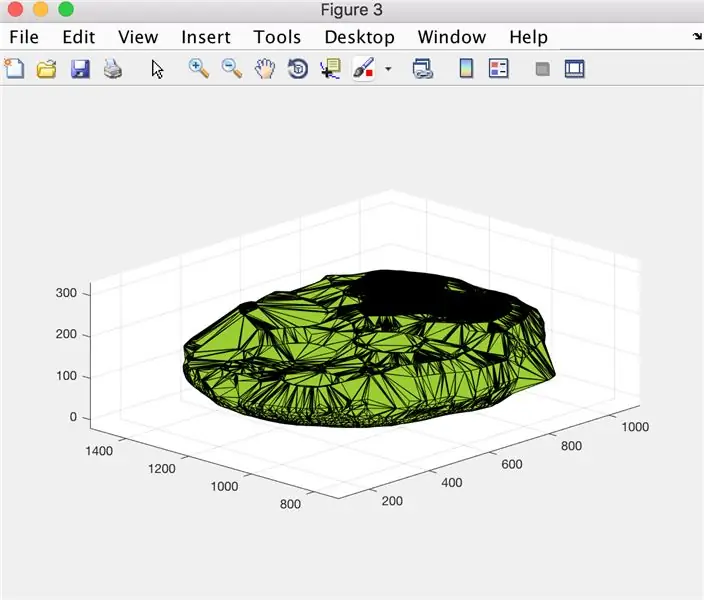
Étape 12: Enregistrement des données: affichage d'un graphique du volume cérébral au fil du temps

- Convertir le tableau de valeurs x (xVals) en un tableau catégoriel (xValsCategorical), pour permettre le traçage
- Générer une figure fenêtre 5
- Tracez les points désignés par "xValsCategorical" et "yVals" (contenant le volume du cerveau), en utilisant des cercles creux pour indiquer les points et être reliés par des lignes pointillées
- Intitulez le tracé comme suit: nom du patient Données sur le volume cérébral
- Étiquetez l'axe des x comme indiqué sur l'image
- Étiquetez l'axe des y comme indiqué sur l'image
- Soit le chiffre 5 égal à la sortie
À partir de là, la fonction patientName appelée produira un fichier avec des données modifiées gardant une trace du volume cérébral au fil du temps, et un graphique affichant les tendances.
Étape 13: Combler les lacunes dans les sous-parcelles: Subplotclose.m

La fonction, adaptée du code de https://www.briandalessandro.com, fonctionne pour combler les écarts entre les figures de sous-intrigue du code principal, lorsque les figures affichant les images IRM et les couches cérébrales sont créées. La fonction de sous-parcelle utilisée dans subplotclose.m ajuste la position des sous-parcelles données pour qu'elles s'ajustent parfaitement les unes aux autres dans l'aspect de la dimension la plus longue. Par exemple, si le code prévoit une matrice 7 x 3, les lignes seront ajustées car la dimension de la ligne est plus longue. Si le code prévoit une matrice 3 x 7, les colonnes seront bien ajustées, avec des espaces dans les lignes, comme indiqué dans les figures de notre code principal.
Étape 14: Le code principal: Tout effacer et demander des entrées

Pour démarrer le code principal, cliquez sur le même bouton qui dit "Nouveau" dans le coin supérieur gauche de la fenêtre, et sélectionnez "Script" au lieu de "Fonction" dans les sections précédentes. Tapez le code comme indiqué dans l'image dans la fenêtre de l'éditeur. Les lignes de code effectuent les tâches suivantes dans l'ordre:
- Fermez tous les fichiers ouverts à l'exception de 0, 1 et 2.
- Fermez toutes les fenêtres de figures.
- Effacez toutes les variables dans l'espace de travail.
- Effacez la fenêtre de commande.
- Affichage dans la fenêtre de commande: veuillez saisir les dimensions suivantes pour les examens IRM:
- Sur une nouvelle ligne dans la fenêtre de commande, demandez: Longueur d'un balayage en millimètres:. La réponse saisie par l'utilisateur sera définie sur la variable "lengthMM".
- Sur une nouvelle ligne, demandez: Distance entre les IRM en millimètres:. La réponse saisie par l'utilisateur sera définie sur la variable "ZStacks".
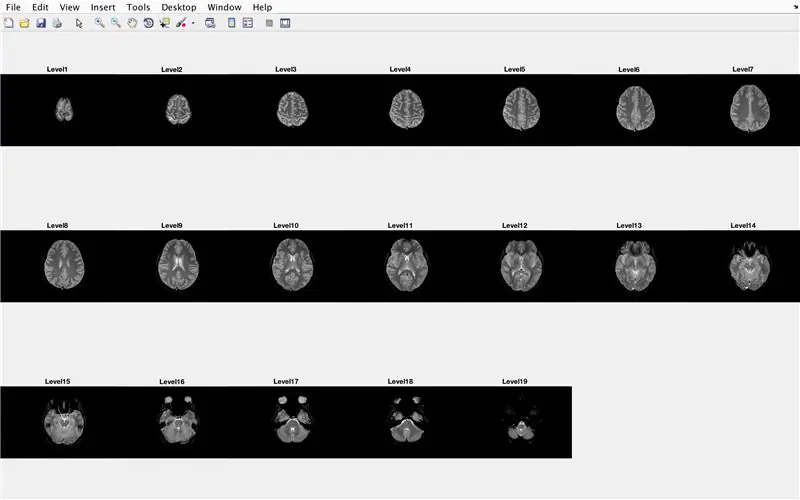
Étape 15: Le code principal: traitement par lots des images

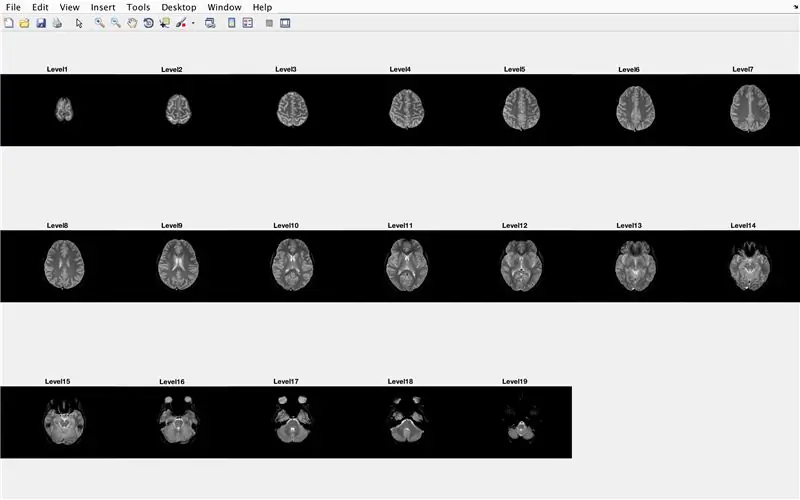
Dans cette section, le code chargera les images (constituées des scans IRM des coupes transversales du cerveau) et stockera les noms de chaque fichier image dans la variable "Base" et affichera chacun des scans IRM. Veuillez suivre avec le code dans l'image, qui fait ce qui suit:
- Créez un tableau de structure "BrainImages" qui contient des informations sur tous les fichiers du dossier actuel qui correspondent au format de nom de MRI_().png
- Définir la variable "NumberofImages" égale au nombre d'éléments dans le tableau de structure "BrainImages"
- Ouvrir la fenêtre de la figure 1
- Définir une boucle for pour parcourir le nombre d'images comptées dans le fichier
- Pour chaque boucle, "CurrentImage" est le nom respectif de chaque fichier MRI_i.png, avec le numéro d'itération comme 'i'
- Générer la sous-intrigue 3 x 7 pour afficher les 19 images à charger par "imshow"
- Afficher chaque image comme un autre élément dans la fenêtre de la figure de sous-intrigue
- Intitulez chaque élément de sous-intrigue comme Level_, où vide est le numéro d'itération de la boucle for.
- Terminer la boucle for (éviter les erreurs)
Cela affichera dans la fenêtre de la figure 1 tous les scans IRM sous forme brute dans une configuration 3 x 7 sans espace dans l'orientation x.
Étape 16: Le code principal: le rembourrage

Avec le remplissage, nous évitons le problème de légères différences dans les tailles d'image qui peuvent entraîner une erreur de non-concordance des dimensions dans le cas où une image est légèrement plus grande qu'une autre.
- Ouvrir la fenêtre de figurine 2
- Chargez la matrice d'image de MRI_1-p.webp" />
- Trouvez la taille de la matrice de l'image et définissez-la sur "OriginalXPixels" (pour le nombre de lignes) et "OriginalYPixels" (pour le nombre de colonnes)
- Configurez la matrice "BrainMat" pour qu'elle se compose de tous les zéros avec 20 lignes supplémentaires et 20 colonnes supplémentaires pour chaque plan, et 19 sections transversales totales, une par plan.
- Configurez "HolesMat" pour qu'il se compose du même tableau tridimensionnel de zéros pour saisir les coordonnées du trou plus tard
- Créez "zeroMat" pour avoir la taille du pavé plus vingt lignes et vingt colonnes, un tableau bidimensionnel de zéros.
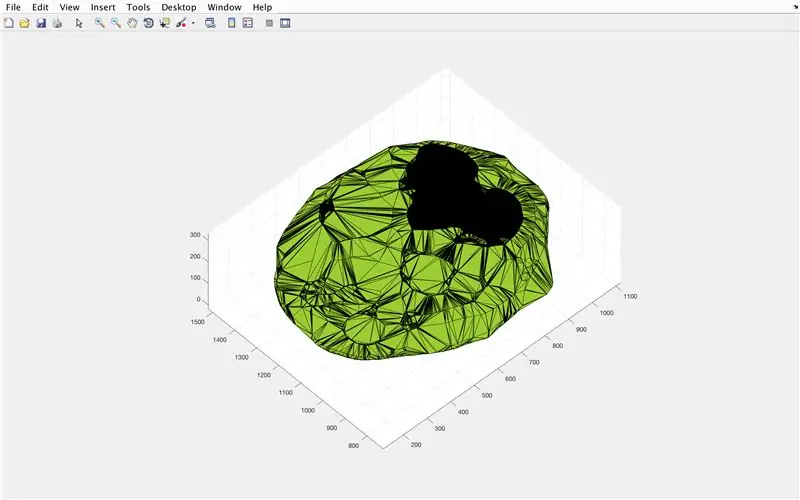
Étape 17: Le code principal: Détermination des limites
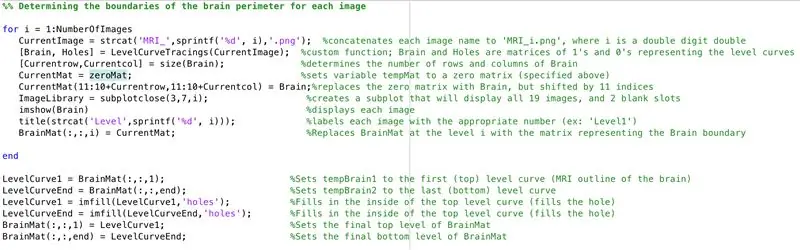
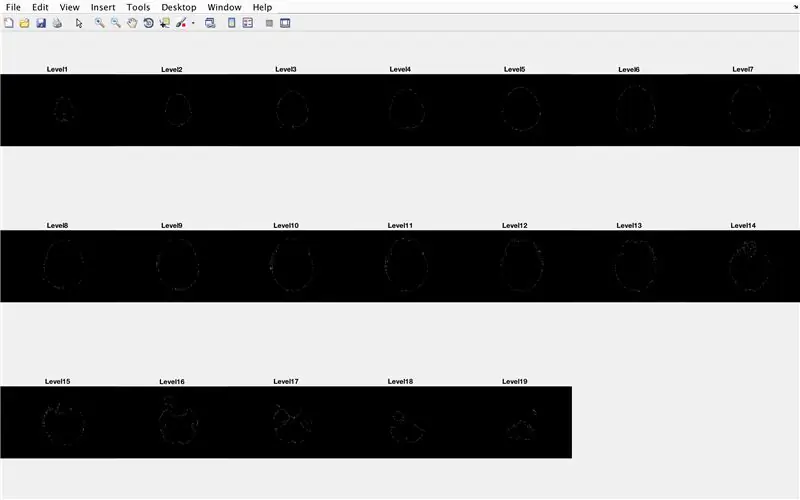
- Définissez une boucle for pour parcourir les données de chaque image chargée précédemment
- De la même manière que le traitement par lots précédemment, "CurrentImage" charge les fichiers avec "MRI_i.png", où i est le numéro d'itération
- Exécutez chaque image via la fonction de traitement "LevelCurveTracings2.m" que vous avez créée précédemment
- Trouvez la taille de la sortie "Brain", définissez le nombre de lignes sur "Currentrow" et le nombre de colonnes sur "Currentcolumns"
- Définissez "CurrentMat" sur une matrice de zéros avec les dimensions spécifiées par "Currentrow" et "Currentcolumns"
- Centrez les données de "Brain" dans "CurrentMat", avec une marge de 10 lignes de tous les côtés
- Générer un sous-parcelle de dimensions 3 x 7, pour afficher les limites des images
- Titre de chacun des éléments de la sous-intrigue dans la fenêtre de la figure
- Générer une matrice tridimensionnelle "BrainMat" composée de chaque couche de limites "CurrentMat"
- Terminer la boucle for (pour éviter les erreurs)
La sous-section suivante remplit les trous laissés en haut et en bas de la forme tridimensionnelle proposée
- Définissez "LevelCurve1" égal à la première couche de "BrainMat" (en bas du solide)
- Définissez "LevelCurveEnd" égal à la couche finale de "BrainMat" (en haut du solide)
- Écraser "LevelCurve1" avec un calque rempli
- Écraser "LevelCurveEnd" avec un calque rempli
- Définissez la couche remplie comme couche inférieure de "BrainMat"
- Définissez la couche remplie comme couche supérieure de "BrainMat"
Étape 18: Le code principal: déterminer la bonne dimension Z

Les trois premières lignes consistent à configurer un tableau vide "z" et à effectuer des opérations de conversion simples (diviser les pixels par longueur) pour obtenir une lecture correcte du volume en mm^3
- Créer une boucle for pour parcourir chaque couche
- Trouver le nombre de uns dans une couche donnée
- Convertissez les coordonnées z pour celles en valeurs qui sont mises à l'échelle au bon rapport, définies sur "tempz", un vecteur de colonne
- Ajouter la valeur z pour la courbe de niveau au vecteur z
Avec cela, les coordonnées z sont ajustées correctement.
Étape 19: Le code principal: Détermination des coordonnées X et Y

Maintenant, déterminez les positions x et y de chacun des points dans les limites.
- Initialiser "xBrain" en tant que tableau vide
- Initialiser "yBrain" en tant que tableau vide
- Configurer une boucle for pour parcourir chaque image chargée
- Compilez une matrice à deux colonnes pour stocker les coordonnées planes de chaque point de la limite, représentées par les vecteurs colonnes "RowBrain" et "ColumnBrain"
- Ajouter "xBrain" avec les coordonnées "RowBrain" actuellement trouvées
- Ajoutez "yBrain" avec les coordonnées "ColumnBrain" actuellement trouvées
- Terminer la boucle for (pour éviter les erreurs)
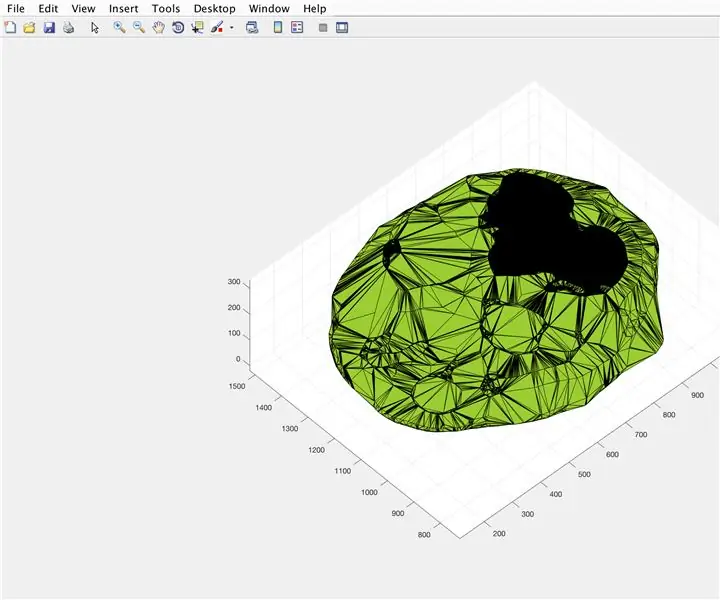
Étape 20: Le code principal: tracer une structure tridimensionnelle, trouver un volume et enregistrer des données
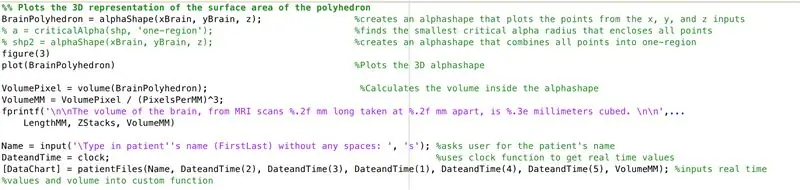

En utilisant la fonction alphaShape, nous allons créer une structure tridimensionnelle à partir de laquelle nous pouvons calculer le volume du cerveau.
- Utilisez la fonction alphaShape, branchez les vecteurs "xBrain", "yBrain" et "z" pour les coordonnées x, y et z, et définissez-les sur "BrainPolyhedron"
- Ouvrir la fenêtre de la figure 3
- Tracer la forme alpha calculée "BrainPolyhedron", afficher dans la fenêtre de la figure
- Calculer le volume de la forme alpha, en utilisant une fonction "volume" qui fonctionne pour les formes alpha
- Convertir le volume en mm^3
- Imprimer le volume du solide dans la fenêtre de commande
- Demander un nom de patient à définir comme entrée
- Obtenez la date et l'heure actuelles avec l'horloge et réglez sur "DateandTime"
- Appeler la fonction "patientFiles" pour enregistrer et tracer les données calculées
À partir de là, les deuxième et troisième images devraient montrer les chiffres qui s'affichent et la quatrième image celle qui devrait être affichée dans la fenêtre de commande.
Conseillé:
Kits de voiture de suivi de robot intelligent bricolage Suivi de voiture photosensible: 7 étapes

DIY Smart Robot Tracking Car Kits Tracking Car Photosensible: Design by SINONING ROBOTVous pouvez acheter à partir de la puce de suivi du robot carTheoryLM393 comparer les deux photorésistances, lorsqu'il y a une LED de photorésistance latérale sur BLANC le côté du moteur s'arrêtera immédiatement, l'autre côté du moteur tourner, de sorte que
Robot Arduino sans fil utilisant le module sans fil HC12 : 7 étapes

Robot Arduino sans fil utilisant le module sans fil HC12 : Salut les gars, bon retour. Dans mon article précédent, j'ai expliqué ce qu'est un circuit de pont en H, un circuit intégré de pilote de moteur L293D, un circuit de pilote de moteur L293D superposé pour piloter des pilotes de moteur à courant élevé et comment vous pouvez concevoir et fabriquer votre propre carte de pilote de moteur L293D
Time Cube - Gadget de suivi du temps Arduino : 6 étapes

Time Cube - Gadget de suivi du temps Arduino: Je voudrais vous proposer un projet arduino simple mais vraiment utile pour suivre les événements temporels en retournant un gadget de cube intelligent. Basculez-le sur "Travail" > "Apprendre" > "Corvées" > "Repos" côté et il comptera le
Suivi GPS en temps réel : 3 étapes
Suivi GPS en temps réel : dans ce didacticiel, nous allons vous montrer comment effectuer un suivi de localisation en temps réel à l'aide d'un BerryGPS-GSM, d'un Raspberry Pi Zero et d'un état initial. Nous enverrons la longitude, la latitude et la vitesse via 3G avec le BerryGPS-GSM à l'état initial
Robot de réseau neuronal Arduino : 21 étapes (avec photos)

Robot de réseau neuronal Arduino : cette instructable est basée sur une série en 3 parties que j'ai réalisée pour la chaîne YouTube Make qui vous montre exactement comment prototyper, concevoir, assembler et programmer votre propre robot de réseau neuronal Arduino. Après avoir regardé la série complète, vous devriez avoir une meilleure
