Table des matières:
- Auteur John Day day@howwhatproduce.com.
- Public 2024-01-30 09:05.
- Dernière modifié 2025-01-23 14:45.
Par: Phuc Lam, Paul Yeung, Eric Reyes
Reconnaître que les erreurs dans la segmentation des poumons produiront de fausses informations concernant l'identification d'une zone pathologique et peuvent affecter directement le processus de diagnostic. Les techniques modernes d'aide informatique n'ont pas réussi à fournir des résultats précis lorsque les maladies pulmonaires ont des formes difficiles. Ces formes anormales peuvent être causées par des épanchements pleuraux, des consolidations, etc. En appliquant la technique de segmentation pulmonaire, dans laquelle les limites du poumon sont isolées du tissu thoracique environnant, notre application peut identifier les limites avec les seuils de saisie de l'utilisateur pour donner des vues entièrement personnalisables des formes des poumons, Le but de ce projet MatLab est de créer une application de segmentation pulmonaire interactive conviviale pour détecter les conditions pathologiques des images radiographiques des poumons. Notre objectif est de créer un moyen plus efficace d'illustrer et d'identifier les poumons anormaux afin de donner aux médecins et aux radiologues un moyen plus fiable de diagnostiquer les maladies pulmonaires. À l'aide de l'outil de conception d'applications de MatLab, le programme est conçu pour fonctionner spécifiquement avec les radiographies pulmonaires et la tomodensitométrie (TDM), mais il est également testé pour fonctionner avec les IRM.
Les instructions ci-dessous contiennent notre technique de filtrage du bruit (filtre de Wiener passe-bas) ainsi que le seuil d'image (en utilisant l'histogramme d'intensité de l'image en niveaux de gris) et en utilisant un gradient morphologique (la différence entre la dilatation et l'érosion d'une image) pour identifier une région d'intérêt. L'instruction expliquera ensuite comment nous intégrons tous les éléments dans l'interface utilisateur graphique (GUI).
Noter:
1). Ce projet s'inspire d'un article de recherche: "Segmentation et analyse d'images de poumons anormaux au scanner: approches actuelles, défis et tendances futures". Qui peut être trouvé ici
2). Nous utilisons des images radiographiques du NIH: Clinical Center. Le lien peut être trouvé ici
3). L'aide du concepteur d'applications peut être trouvée ici
4). Avant d'exécuter le code: vous devez changer le chemin Dir (en ligne 34) vers votre répertoire de fichiers et le type d'image (ligne 35) (nous analysons *.png).
Étape 1: Étape 1: Chargement de l'image
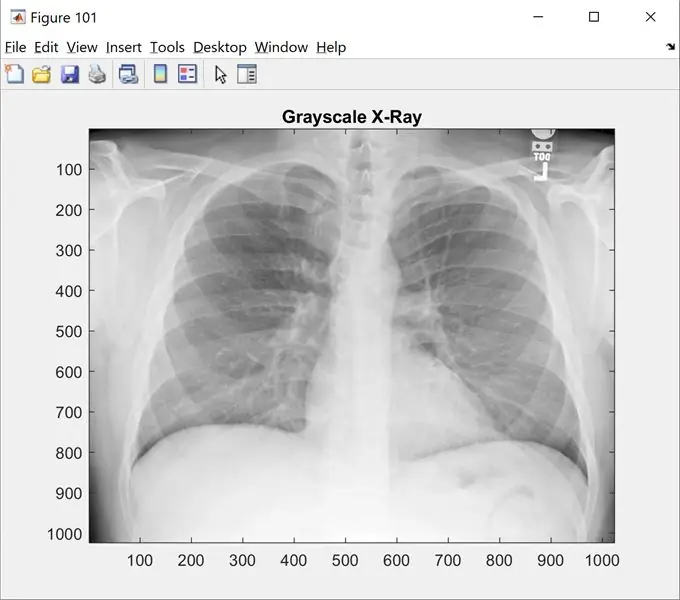
Cette étape vous montrera l'image originale en niveaux de gris. Remplacez le 'name_of_picture.png' par le nom de votre image
dégager; clc; ferme tout;
%% Chargement des images
raw_x_ray='name_of_picture.png';
I=imread(raw_x_ray);
chiffre(101);
imshow(I);
palette de couleurs (gris);
title('Rayon X en niveaux de gris');
Étape 2: Étape 2: Filtrage du bruit et histogramme
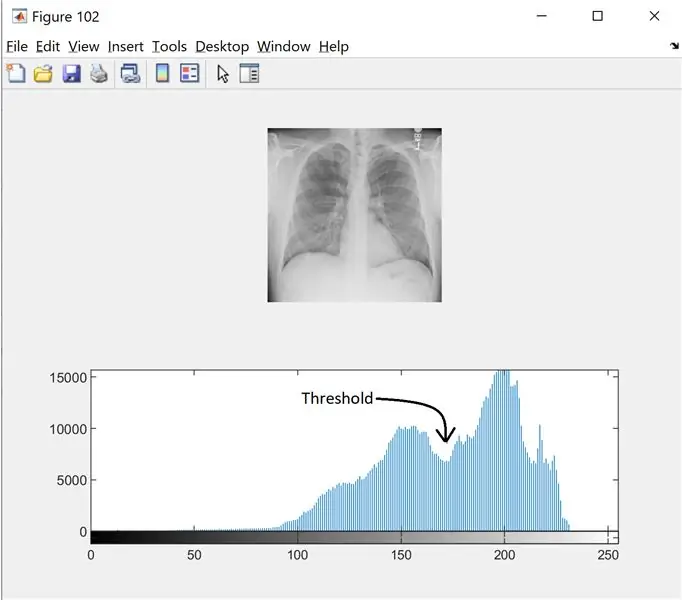
Afin de trouver le seuil pour l'image en niveaux de gris, nous regardons l'histogramme pour voir s'il existe des modes distincts. Lire la suite ici
I=wiener2(I, [5 5]);
chiffre(102);
sous-intrigue(2, 1, 1);
imshow(I);
sous-intrigue(2, 1, 2);
imhist(I, 256);
Étape 3: Étape 3: Définition des seuils
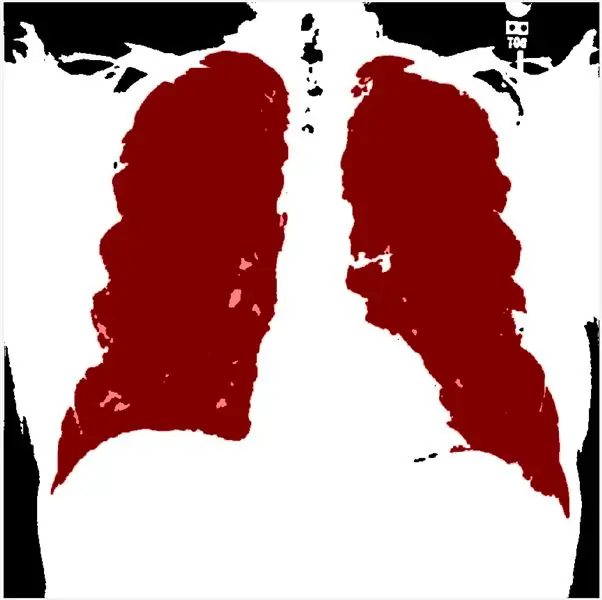
Cette étape permet de définir le seuil en fonction de l'histogramme. morphologicalGradient mettra en évidence la région d'intérêt en rouge, et la fonction visboundaries superpose l'image esquissée et filtrée du poumon en rouge.
En utilisant regionprops, nous pouvons déterminer les tableaux de solidité et les trier par ordre décroissant. Ensuite, je binarise l'image sclae grise et applique la méthode du gradient morphologique et mLoren Shurasking pour mettre en évidence la région d'intérêt (ROI). L'étape suivante consiste à inverser l'image pour que la ROI du poumon soit blanche sur fond noir. J'utilise la fonction showMaskAsOverlay pour afficher 2 masques. Remarque: le code est inspiré de Loren Shure, lien.
Enfin, je crée un contour rouge en utilisant les bwbwboundaries et en masquant l'image du filtre et les limites.
a_seuil = I >= 172; % définir ce seuil
[labelImage, numberOfBlobs] = bwlabel(a_thresh);
props = regionprops(a_thresh, 'all');
triéSolidity = sort([props. Solidity], 'descendre');
SB = triéSolidité(1);
si SB == 1 % SB n'accepter que la solidité == 1 filtrer les os
imagebinaire = imbinarize(I); chiffre(103);
imshow(imagebinaire); palette de couleurs (gris);
SE = strel('carré', 3);
morphologicalGradient = imsubtract(imdilate(binaryImage, SE), imerode(binaryImage, SE));
mask = imbinarize(morphologicalGradient, 0.03);
SE = strel('carré', 2);
masque = imclose(masque, SE);
mask = imfill(masque, 'trous');
masque = bwareafilt(masque, 2); % numéro de contrôle de la zone d'exposition
notMask = ~masque;
masque = masque | bwpropfilt(pasMask, 'Zone', [-Inf, 5000 - eps(5000)]);
showMaskAsOverlay(0.5, masque, 'r'); % vous devez télécharger l'application/la fonction showMaskAsOverlay
BW2 = imfill(binaryImage, 'holes');
nouvelle_image = BW2;
nouvelle_image(~masque) = 0; % fond et trous inversés
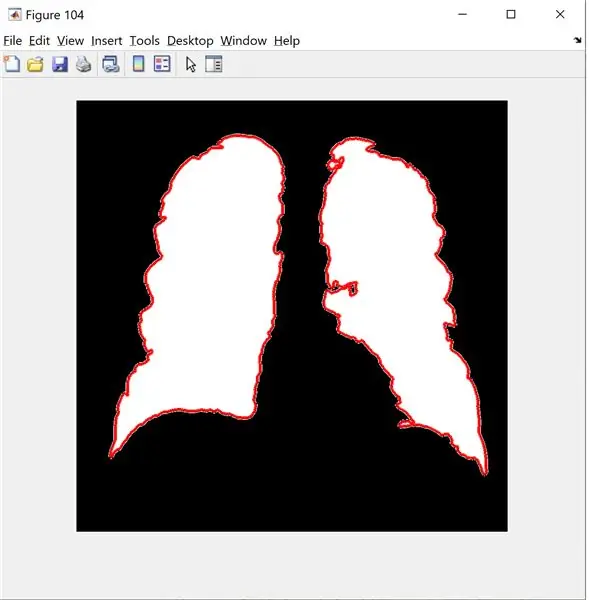
B=bwboundaries(new_image); % ne peut accepter que 2 dimensions
chiffre(104);
imshow(nouvelle_image);
attendez
visfrontières(B);
finir
Étape 4: Création de l'interface graphique
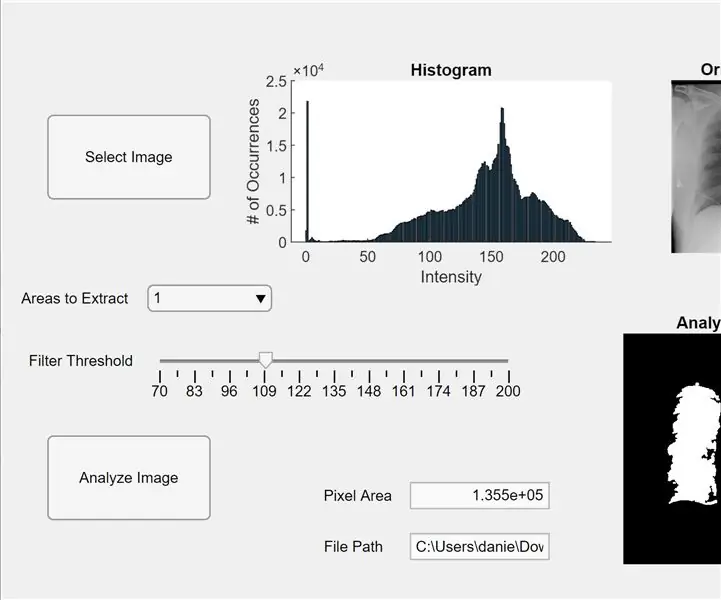
Maintenant, nous intégrons le code précédent dans une application MATLAB. Ouvrez l'App Designer dans MATLAB (Nouveau > App). Tout d'abord, nous concevons l'interface en cliquant-maintenant et en faisant glisser sur trois axes dans l'espace de travail central. Ensuite, nous cliquons et faisons glisser deux boutons, un champ d'édition (texte), un champ d'édition (numérique), un curseur et un menu déroulant. Deux axes afficheront chacun l'aperçu et l'analyse de l'image, et le troisième axe affichera un histogramme de pixels pour l'aperçu de l'image « sélectionnée ». Le champ d'édition (texte) affichera le chemin du fichier de l'image sélectionnée, et le champ d'édition (numérique) affichera la zone de pixels détectée des poumons.
Passez maintenant du mode conception au mode code dans App Designer. Entrez dans le code le code des propriétés en cliquant sur le bouton rouge « Propriétés » avec un signe plus à côté. Initialisez les propriétés I, seuil et regionsToExtract comme dans le code fourni ci-dessous. Ensuite, cliquez avec le bouton droit sur un bouton dans le coin supérieur droit de l'espace de travail (le navigateur de composants) et accédez à Rappels> Aller à… rappel. Ajoutez le code pour « fonction SelectImageButtonPushed(app, event). » Ce code vous permet de sélectionner une image à analyser depuis votre ordinateur à l'aide de uigetfile. Après avoir sélectionné une image, une image de prévisualisation apparaîtra sous les axes accompagnée d'un histogramme. Ensuite, faites un clic droit sur l'autre bouton et répétez la même procédure pour créer une fonction de rappel.
Ajoutez le code sous « function AnalyzeImageButtonPushed (app, event). » Ce code effectuera le comptage de pixels et la détection de gouttes sur l'image d'aperçu sur le bouton d'analyse de l'image (celui sur lequel vous avez cliqué avec le bouton droit pour ce code). Après avoir programmé les boutons, nous allons maintenant programmer le curseur et le menu déroulant. Cliquez avec le bouton droit sur le curseur, créez une fonction de rappel et ajoutez le code sous « fonction FilterThresholdSliderValueChanged(app, event) » jusqu'à la fin. Cela permet au curseur d'ajuster le seuil d'intensité des gris.
Créez une fonction de rappel pour le menu déroulant et ajoutez le code sous « fonction AreastoExtractDropDownValueChanged(app, event) » pour permettre au menu déroulant de modifier le nombre de blobs affichés sur les axes de l'image analysée. Maintenant, cliquez sur chaque entité dans le navigateur de composants et modifiez leurs propriétés à votre guise, telles que la modification des noms des entités, la suppression des axes et la modification de la mise à l'échelle. Faites glisser et déposez les entités du navigateur de composants dans la vue Conception vers une disposition fonctionnelle et facile à comprendre. Vous avez maintenant une application dans MATLAB qui peut analyser des images de poumons pour la zone de pixels !
propriétés (Accès = privé)I = ; % fichier d'image
seuil = 257; % seuil pour la binarisation de l'intensité des gris
régionsVersExtraire = 2;
finir
fonction SelectImageButtonPushed (application, événement)
clc;Dir = 'C:\Users\danie\Downloads\images_004\images'; %define invariable fichier "préfixe"
[imageExt, chemin] = uigetfile('*.png'); % récupérez la partie variable du nom de l'image
imageName = [Dir filesep imageExt]; %concaténer les souches invariables et variables
app. I = imread(imageName); %lire l'image
imshow(app. I, 'parent', app. UIAxes); %afficher l'image
app. FilePathEditField. Value = chemin; % afficher le chemin du fichier d'où provient l'image d'origine
finir
fonction AnalyzeImageButtonPushed (application, événement)
image d'origine = app. I;
originalImage = wiener2(app. I, [5 5]); % filtre de suppression de points
histogramme (app. AxesHistogram, app. I, 256); % afficher l'histogramme de l'image
a_thresh = originalImage >= app.threshold; % définir ce seuil
labelImage = bwlabel(a_thresh);
props = regionprops(a_thresh, 'all');
triéSolidity = sort([props. Solidity], 'descendre');
SB = triéSolidité(1);
si SB == 1 % SB n'accepter que la solidité ==1 filtrer les os
SE = strel('carré', 3);
morphologicalGradient = imsubtract(imdilate(labelImage, SE), imerode(labelImage, SE));
mask = imbinarize(morphologicalGradient, 0.03);
SE = strel('carré', 2);
masque = imclose(masque, SE);
mask = imfill(masque, 'trous');
mask = bwareafilt(mask, app.regionsToExtract);
% numéro de contrôle de la zone d'exposition
notMask = ~masque;
masque = masque | bwpropfilt(pasMask, 'Zone', [-Inf, 5000 - eps(5000)]);
BW2 = imfill(labelImage, 'trous');
nouvelle_image = BW2;
nouvelle_image(~masque) = 0;
B = bwboundaries(new_image); % ne peut accepter que 2 dimensions imshow(new_image, 'parent', app. UIAxes2);
hold(app. UIAxes2, 'on');
visfrontières(B);
set(gca, 'YDir', 'reverse');
zone poumon = bwarea(nouvelle_image);
app. PixelAreaEditField. Value = lungArea;
finir
finir
fonction FilterThresholdSliderValueChanged (application, événement)
app.threshold = app. FilterThresholdSlider. Value;
finir
function AreastoExtractDropDownValueChanged(app, event)stringNumber = app. AreatoExtractDropDown. Value;
app.regionsToExtract = str2double(stringNumber);
finir
finir
Conseillé:
Détection facile des visages MATLAB : 4 étapes

MATLAB Easy Face Detection: L'objectif principal de ces instructables est de montrer à quel point le traitement de l'image sera facile. Avec l'aide de MATLAB, la détection et le suivi des visages ont été un domaine de recherche important et actif, c'est pourquoi je vais expliquer comment cela peut être fait avec
Comment coder et publier Matlab 2016b vers Word (Guide du débutant): 4 étapes

Comment coder et publier Matlab 2016b dans Word (Guide du débutant) : Matlab est un programme de langage hautes performances utilisé pour calculer les résultats techniques. Il a la capacité d'intégrer des visuels, des calculs et une programmation de manière conviviale. Avec ce programme, l'utilisateur peut publier des problèmes et des solutions
Contrôler Raspberry Pi à l'aide de Matlab : 5 étapes

Contrôler Raspberry Pi à l'aide de Matlab : Hé, ce tutoriel concerne le contrôle de votre raspberry pi sans tête à l'aide de matlab. Vous devrez peut-être installer matlab sur la dernière version pour prendre en charge la nouvelle carte Raspberry Pi
Contrôleur robotique ROS basé sur Matlab : 9 étapes
Contrôleur robotique ROS basé sur Matlab : depuis que je suis enfant, j'ai toujours rêvé d'être Iron Man et je le fais toujours. Iron Man est l'un de ces personnages qui est réalistement possible et tout simplement j'aspire à devenir Iron Man un jour même si les gens se moquent de moi ou disent que c'est impossible
Améliorations de la programmation d'entraînement dans MATLAB : 6 étapes

Améliorations de la programmation des trains dans MATLAB : objectif : l'objectif de ce système programmé est d'examiner un Arduino à petite échelle et d'appliquer le codage à plus grande échelle pour potentiellement améliorer certaines fonctionnalités de sécurité des systèmes Amtrak Railroad. Pour ce faire, nous avons ajouté un capteur d'humidité du sol
