Table des matières:
- Auteur John Day day@howwhatproduce.com.
- Public 2024-01-30 09:08.
- Dernière modifié 2025-01-23 14:46.
Par: Madhumita Kannan, Henry Nguyen, Ashley Urrutia Avila, Mei Jin
Ce code MATLAB est un programme permettant de détecter la taille, la forme et l'emplacement exacts d'une tumeur trouvée dans les IRM cérébrales d'un patient. Ce programme est conçu pour fonctionner à l'origine avec la détection de tumeurs dans les scanners IRM du cerveau, mais il peut également être utilisé pour le diagnostic du cancer dans d'autres scanners d'organes.
Les instructions suivantes décriront d'abord les méthodes d'analyse d'image par filtrage et nettoyage de l'IRM, par binarisation, filtrage médian et fenêtres coulissantes. Ensuite, il expliquera comment isoler la tumeur à l'aide d'un masque elliptique pré-généré et le filtrer davantage pour délimiter le périmètre de la forme de la tumeur.
Une fois la tumeur détectée, les instructions décriront plus en détail comment incorporer ce programme dans une interface utilisateur graphique (GUI). Tout au long de ces instructions, le code et les fichiers appropriés seront joints pour aider à expliquer le fonctionnement de cette analyse par IRM.
Certaines choses à savoir, à télécharger et à préparer avant de procéder avec cette instructable:1. Assurez-vous d'avoir téléchargé la dernière version de MATLAB. Vous pouvez installer R2018b ici:
2. Pour exécuter ce programme, vous devez avoir accès aux fichiers d'IRM cérébrale. Bien que certaines puissent toujours être trouvées à partir d'images Google, une analyse approfondie et précise peut être effectuée à partir d'images appropriées de différentes couches de scans cérébraux pour chaque patient. Vous pouvez accéder aux fichiers de 20 patients différents atteints de glioblastome avant et après traitement à partir de cette base de données:
3. L'objectif de ce programme et les différentes méthodes qui guident ce projet sont décrits dans ce document de recherche:
Étape 1: Lancer l'interface utilisateur graphique (GUI)
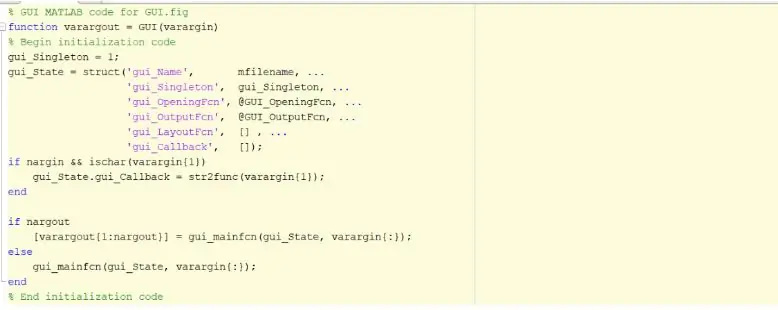
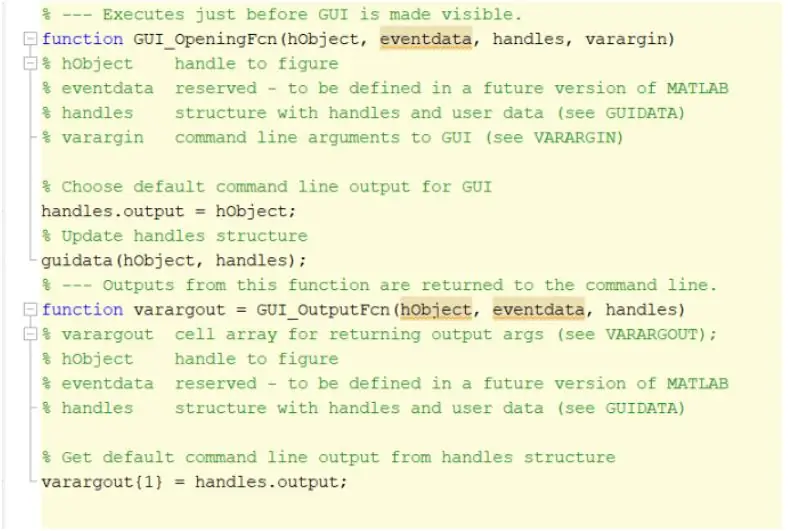
La première étape serait de créer et d'initier l'interface utilisateur graphique, GUI. Cela peut être fait en tapant guide dans la fenêtre de commande, en appuyant sur Entrée et en créant une nouvelle interface graphique. Une fois cette étape terminée, vous pouvez commencer à créer des fonctions telles que des axes, du texte statique, du texte d'édition et des boutons-poussoirs qui seront affichés une fois le programme exécuté et avec lesquels l'utilisateur pourra interagir. Ces fonctions peuvent être modifiées et manipulées via l'inspecteur de propriétés, mais la caractéristique la plus importante qui doit être modifiée lors de la création de ces fonctions est le nom de la balise. Il est important de changer le nom de balise de chaque fonction implémentée car cela nous permettra de créer une fonction de rappel distincte. Une fois que vous êtes satisfait de la disposition de votre interface graphique, vous pouvez passer au chargement des fichiers DICOM qui seront affichés dans l'interface graphique.
Étape 2: Chargement et lecture des images IRM dans MATLAB
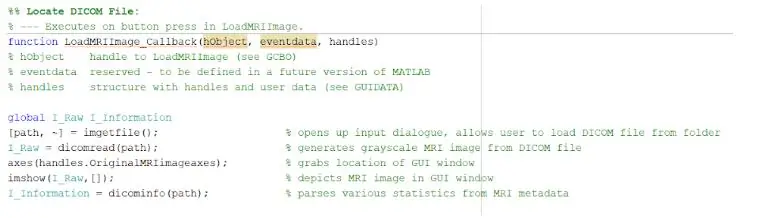
Afin de charger les fichiers DICOM, vous devrez initialiser correctement la fonction de rappel qui serait exécutée en appuyant sur le bouton « Charger l'image IRM ». Une fois cette opération terminée, vous devez créer une variable globale qui afficherait l'image sur les axes des poignées où vous souhaitez que l'image IRM d'origine soit affichée. Les images d'IRM téléchargées à partir de la base de données sont tous des fichiers au format DICOM qui doivent être chargés dans votre répertoire MATLAB. Localisez le fichier en utilisant imgetfile afin de les charger dans le programme. Les images sont lues à l'aide de la fonction MATLAB intégrée « dicomread », et la première image brute de chaque fichier est intégrée dans les axes gauche de l'interface graphique à l'aide d'imshow.
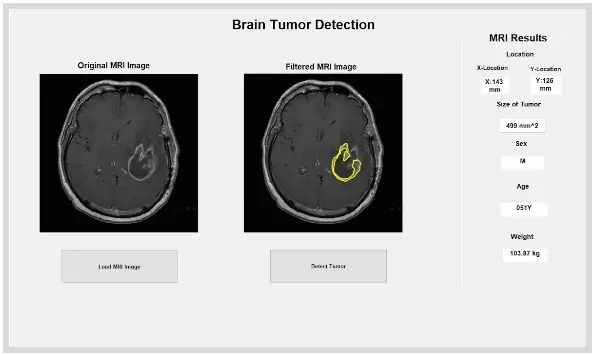
La fonction MATLAB intégrée « dicominfo » est également extrêmement utile pour traiter toutes les informations de chaque fichier IRM dicom. Nous avons utilisé cette fonction pour extraire toutes les informations descriptives des patients, telles que leur sexe, leur âge, leur poids et leur taille. Cette fonction vous fournit également l'ordre de la pile qui est utile pour la mise en œuvre du programme dans l'interface utilisateur graphique. Nous avons créé des variables pour chacune des informations descriptives des patients qui seront utilisées pour l'interface graphique lorsque le bouton de détection est enfoncé.
Étape 3: Filtrage d'images

Une fois le fichier DICOM de l'image brute chargé et lu, l'image doit être convertie des niveaux de gris en une forme binarisée composée uniquement de pixels noirs et blancs. Nous avons utilisé la fonction « imbinariser » pour créer une image binaire à partir de l'image brute en contrôler les aspects du seuillage adaptatif à une valeur de sensibilité de 0,59. Le facteur de sensibilité de seuil par défaut, 0,5, était faible et incapable de détecter les taches et les taches les plus lumineuses de l'image, nous l'avons donc augmenté à 0,59.
L'image binarisée est ensuite traitée à travers un filtre médian à l'aide de la fonction « medfilt2 » car l'image binarisée est bidimensionnelle. Nous définissons chaque pixel de sortie pour contenir la valeur médiane dans le voisinage 5 x 5 autour du pixel correspondant dans l'image binarisée d'entrée. Cela réduit le bruit et préserve les bords dans un carré de 5 x 5 autour de chaque pixel. Ensuite, nous appliquons une fenêtre glissante à l'aide de « strel », pour créer un élément structurant plat en forme de disque avec un rayon de voisinage de 2 pour identifier chaque pixel central d'origine, dans chaque voisinage de disque. Nous avons utilisé un élément structurant de disque car nous analysons chaque tache circulaire et les pixels à l'intérieur de chaque tache, donc un élément en forme de disque est plus utile.
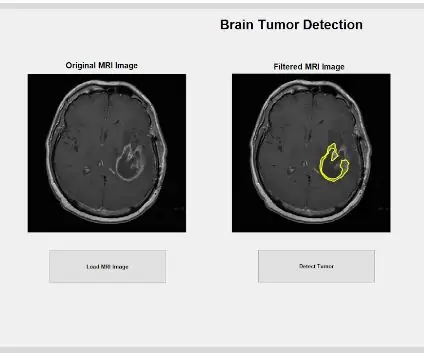
Une fois l'image filtrée, elle peut être nettoyée à l'aide de la fonction « imclose » pour supprimer les points noirs entre les pixels blancs filtrés de l'image et fermer tous les espaces qui l'entourent. L'image complètement traitée peut ensuite être tracée dans le deuxième sous-parcelle de la figure pré-allouée, permettant une comparaison entre l'image brute et filtrée.
Étape 4: Isolement de la tumeur à l'aide d'un masque elliptique


Les points lumineux de la tumeur peuvent ensuite être isolés de l'image filtrée principale à travers un masque elliptique pré-généré. Pour créer ce masque, vous devez connaître la taille de l'image IRM brute d'origine et, en utilisant sa longueur de ligne et de colonne, respectivement comme coordonnées x et y, allouer les coordonnées centrales de l'elliptique. Nous définissons l'axe des y comme un axe majeur avec un rayon de 50 unités du centre et l'axe mineur avec un rayon de 40 unités du centre.
Nous avons utilisé la fonction 'meshgrid' de MATLAB pour générer un plan cartésien avec des coordonnées de grille à deux dimensions basées sur les coordonnées contenues dans les vecteurs de 1 à la longueur de l'axe x, et de 1 à la longueur de l'axe y de l'image. Col est une matrice où chaque ligne est une copie de l'axe des x, et Row est une matrice où chaque colonne est une copie de l'axe des y. La grille cartésienne représentée par les coordonnées Col et Row a des lignes de longueur (1:Y_Size) et des colonnes de longueur (1:X_Size). Utilisez les indices de Col et Row générés par la grille cartésienne pour déterminer l'équation de l'ellipse en fonction du rayon prédéterminé et des coordonnées du centre. Le contour elliptique peut maintenant être rempli avec les pixels blancs trouvés sur les taches tumorales.
En utilisant le masque elliptique pré-généré, nous pouvons recadrer la tumeur spécifique que vous souhaitez analyser à partir de l'image filtrée. Le masque elliptique détecte les taches qui s'intègrent logiquement dans le contour de l'ellipse et accepte cela comme une tache sur l'image filtrée pour être acceptable en tant que tumeur. La fonction « bwareafilt » filtre ensuite tous les autres objets en dehors de cette tumeur détectée de l'image. Nous avons utilisé une fenêtre spécifique de 500 par 4000 empiriquement basée sur les dimensions de toutes les images. Nous avons ensuite appliqué une autre fenêtre coulissante avec « strel » comme élément structurant en forme de disque plat d'un plus grand rayon de voisinage de 6, pour combler les espaces entre chaque pixel blanc central au sein de la tumeur détectée. La tache tumorale détectée est ensuite nettoyée à l'aide de « imclose » pour éliminer davantage les pixels noirs et combler tous les trous avec « imfill ». Cette tumeur traitée peut ensuite être affichée dans le troisième sous-parcelle de la parcelle préallouée pour fournir une comparaison entre la tumeur isolée et les images originales et filtrées de l'IRM.
Étape 5: Décrire la tumeur
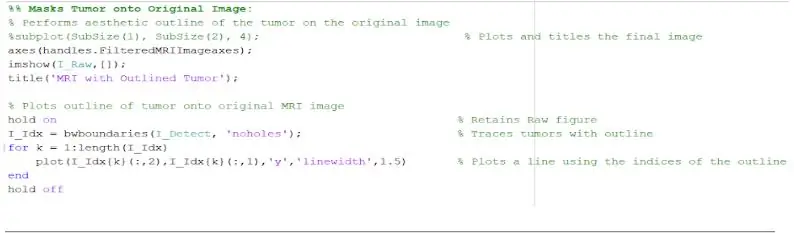
Maintenant que la tumeur est isolée avec le masque, elle peut être délimitée et affichée sur l'image d'origine, pour montrer son emplacement exact. Pour ce faire, nous avons utilisé la fonction « bwboundaries » pour tracer la tumeur précédemment détectée avec un contour. Nous avons spécifié que le contour n'incluait pas les trous dans l'objet tumeur au fur et à mesure de son contour. Cela peut être tracé sur l'image brute d'origine, en utilisant une boucle "for" qui trace le contour autour de la tumeur en utilisant les indices de la ligne avec une largeur de ligne de 1,5 pixel. Ce contour est ensuite tracé sur l'image brute, montrant la taille et l'emplacement exacts de la tumeur, par rapport à l'IRM d'origine.
Étape 6: Analyser les propriétés physiques des tumeurs
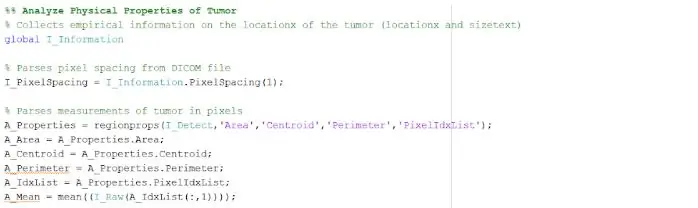
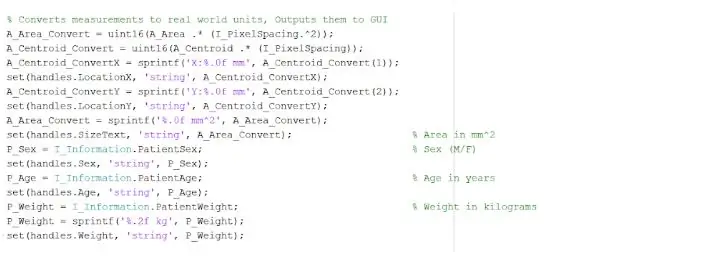
La tache isolée et délimitée peut nous fournir des informations utiles sur la taille, la superficie et l'emplacement de la tumeur. Nous avons utilisé la fonction « regionprops » pour détecter les propriétés de la tumeur qui se rapportent à la zone, au périmètre, aux centroïdes et à la valeur de l'indice de pixel. Cette valeur d'indice de pixel nous donne les unités du monde réel pour chaque pixel de chaque image, uniques à chaque numérisation. Ces propriétés peuvent ensuite être converties en unités réelles de millimètres. Les informations empiriques que le programme nous fournit sont uniques pour chaque IRM et sont extrêmement utiles pour déterminer la taille, l'emplacement et le type de tumeur, que les utilisateurs peuvent analyser et intégrer dans l'interface utilisateur graphique.
Conseillé:
Détection facile des visages MATLAB : 4 étapes

MATLAB Easy Face Detection: L'objectif principal de ces instructables est de montrer à quel point le traitement de l'image sera facile. Avec l'aide de MATLAB, la détection et le suivi des visages ont été un domaine de recherche important et actif, c'est pourquoi je vais expliquer comment cela peut être fait avec
Détection simple des couleurs à l'aide d'OpenCV : 6 étapes
Détection de couleur simple à l'aide d'OpenCV : Salut ! Aujourd'hui, je vais montrer une méthode simple pour détecter une couleur à partir d'une vidéo en direct à l'aide d'OpenCV et de python. Fondamentalement, je vais simplement tester que la couleur requise est présente ou non dans le cadre d'arrière-plan et à l'aide des modules OpenCV, je vais masquer cette région et
Détection des couleurs en Python à l'aide d'OpenCV : 8 étapes
Détection des couleurs en Python à l'aide d'OpenCV : Bonjour ! Cette instructable est utilisée pour expliquer comment extraire une couleur spécifique d'une image en python à l'aide de la bibliothèque openCV. Si vous débutez dans cette technique, ne vous inquiétez pas, à la fin de ce guide, vous pourrez programmer votre propre couleur
Détection d'obstacles RoboCar à l'aide d'un smartphone à l'aide d'Arduino : 5 étapes

Détection d'obstacles RoboCar exploité par smartphone à l'aide d'Arduino : Dans ce projet, nous avons réalisé un Robocar dans lequel deux capteurs à ultrasons, un module Bluetooth a été interfacé avec Arduino
Détection des couleurs à l'aide de LED RVB : 4 étapes

Détection des couleurs à l'aide de LED RVB : Avez-vous déjà voulu un moyen automatisé de détecter la couleur d'un objet ? En projetant une lumière d'une certaine couleur sur l'objet et en regardant la quantité de lumière réfléchie, vous pouvez savoir de quelle couleur est l'objet. Par exemple, si vous allumez un feu rouge o
