Table des matières:
- Étape 1: Prérequis
- Étape 2: Prérequis (suite)
- Étape 3: Prérequis (suite)
- Étape 4: Prérequis (suite)
- Étape 5: Nettoyez Matlab pour préparer l'exécution du code
- Étape 6: sélectionnez 10 images d'œil normal et 10 images présentant des symptômes de rétinopathie diabétique
- Étape 7: sélectionnez 10 images d'œil normal et 10 images présentant des symptômes de rétinopathie diabétique (suite)
- Étape 8: créez 2 variables (normales et diagnostiquées) et définissez-les chacune égales à 0
- Étape 9: créez une boucle for pour télécharger automatiquement des images normales
- Étape 10: créer une boucle for pour télécharger automatiquement des images normales (suite)
- Étape 11: recadrer les bordures de l'image
- Étape 12: Créer une image en niveaux de gris
- Étape 13: Créez une image contrastée
- Étape 14: Améliorer l'image de contraste
- Étape 15: créer un filtre moyen
- Étape 16: Combinez le filtre de moyenne avec l'image contrastée
- Étape 17: Créer un nouveau masque moyen en soustrayant des pixels
- Étape 18: créer une image filtrée binaire
- Étape 19: Supprimer les petits blobs trouvés dans les images filtrées
- Étape 20: Créer un élément de structuration de disque
- Étape 21: Effectuer des opérations de fermeture morphologique
- Étape 22: Trouvez les objets avec une connectivité d'au moins 8
- Étape 23: Trouvez le nombre maximum de pixels connectés
- Étape 24: définissez les valeurs de pixels maximales sur 0 et recherchez les pixels avec une connectivité >=26 pixels
- Étape 25: Supprimer les vaisseaux sanguins dans l'image
- Étape 26: Affichage des chiffres
- Étape 27: Retirez les vaisseaux et comptez les gouttes de sang
- Étape 28: Diagnostiquer l'image rétinienne en fonction du nombre de caillots sanguins identifiés
- Étape 29: S'il y a plus de 5 blobs…
- Étape 30: Répétez le processus de filtrage pour les images normales avec des valeurs de chiffre d'image comme 2 et 3
- Étape 31: Répétez le processus entier pour les images diagnostiquées
- Étape 32: Analyse statistique
- Étape 33: Trouver l'intervalle de confiance
- Auteur John Day day@howwhatproduce.com.
- Public 2024-01-30 09:09.
- Dernière modifié 2025-01-23 14:46.

(Voir le contour du code ci-dessus)
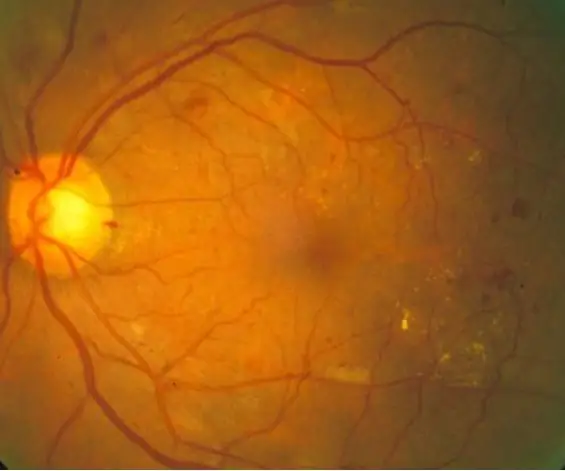
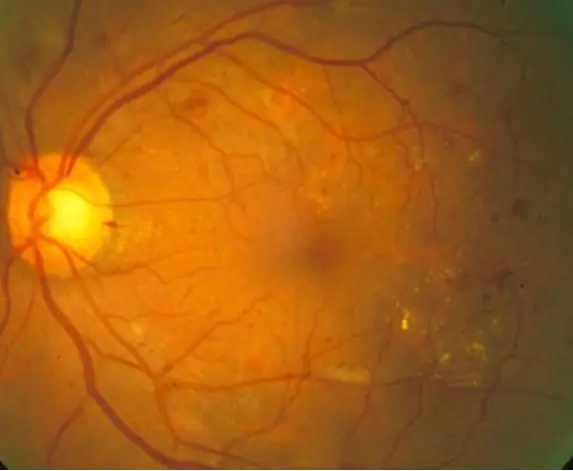
La rétinopathie diabétique est une maladie oculaire liée au diabète causée par une glycémie élevée. Les niveaux élevés de sucre dans le sang font gonfler les vaisseaux sanguins de la rétine, ce qui entraîne une hypertrophie des vaisseaux sanguins et même des fuites de vaisseaux, ce qui entraîne des taches sombres sur les images rétiniennes. Avec ce code, nous visons à utiliser l'apparition de points de fuite dans les vaisseaux sanguins comme indicateur de rétinopathie diabétique de fond, bien que des techniques de diagnostic supplémentaires soient nécessaires dans le monde réel. L'objectif de ce code est d'automatiser le traitement des images et le diagnostic des images rétiniennes pour identifier les signes de rétinopathie diabétique montrés par des taches sombres dans les images rétiniennes.
10 images rétiniennes normales et 10 images rétiniennes diagnostiquées ont été traitées via un code qui lit et filtre d'abord les images, puis quantifie les taches sombres pour déterminer si des symptômes de rétinopathie diabétique sont présents, sur la base d'un seuil donné. Les résultats sont ensuite imprimés sur la fenêtre de commande pour l'interprétation du spectateur.
Étape 1: Prérequis
1. Assurez-vous que le programme MATLAB est téléchargé sur votre ordinateur.
2. Téléchargez le fichier txt trouvé dans le lien. (Appuyez sur 'ctrl + s' pour enregistrer dans le même répertoire que le code MATLAB)
Étape 2: Prérequis (suite)
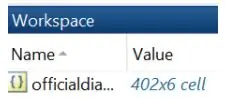
4. Ouvrez MATLAB et tapez « uiimport » dans la fenêtre de commande.
5. Sélectionnez le fichier officialdiagnoses.txt et importez-le dans MATLAB en tant que matrice de cellules.
6. Assurez-vous de voir « diagnostics officiels » en tant que variable dans l'espace de travail.
Étape 3: Prérequis (suite)

7. Téléchargez la fonction ModWald.m, qui peut être obtenue à partir du code ci-dessus ou téléchargez-la à partir de Canvas.
(Code fourni par le professeur King et le professeur Choi)
Étape 4: Prérequis (suite)

8. Téléchargez les 400 images brutes de la section des données du projet STARE.
Étape 5: Nettoyez Matlab pour préparer l'exécution du code

Ajouter au code:
1. tout fermer (ferme toutes les images précédemment ouvertes)
2. clearvars - except officialdiagnoses (efface toutes les variables à l'exception du fichier txt de diagnostics officiels précédemment importé)
3. cclc (efface la fenêtre de commande)
Étape 6: sélectionnez 10 images d'œil normal et 10 images présentant des symptômes de rétinopathie diabétique
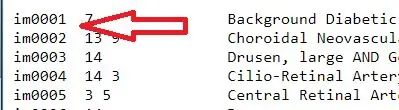
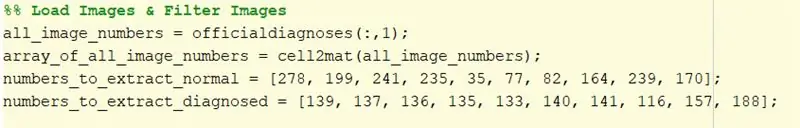
1. Prenez le fichier texte de diagnostic et extrayez les noms des images. Ces noms sont contenus dans la première colonne du fichier texte donc pour les extraire tapez 'officialdiagnoses(:, 1)'. La matrice des noms d'images a été affectée à une variable, « all_image_numbers »
2. Convertissez la variable all_image_numbers d'un tableau de cellules en un tableau matriciel à l'aide de la fonction cell2mat
Étape 7: sélectionnez 10 images d'œil normal et 10 images présentant des symptômes de rétinopathie diabétique (suite)
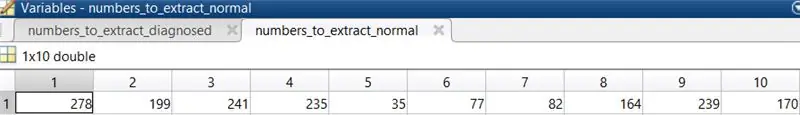
3. Sélectionnez 10 images oculaires normales pour exécuter le code. Les images sélectionnées dans ce cas étaient 278, 199, 241, 235, 35, 77, 82, 164, 239, 170.
Placez ces nombres dans une matrice et affectez-les à une variable qui sera appelée lors du chargement des images.
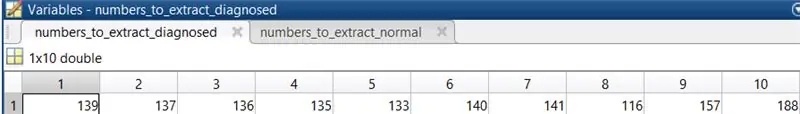
4. Répétez l'étape 3 pour les images rétiniennes qui ont été diagnostiquées avec une rétinopathie diabétique. Les images sélectionnées dans ce cas étaient 139, 137, 136, 135, 133, 140, 141, 116, 157, 188.
Étape 8: créez 2 variables (normales et diagnostiquées) et définissez-les chacune égales à 0

Créez ces variables avant la boucle for pour initialiser les numéros de boucle.
Étape 9: créez une boucle for pour télécharger automatiquement des images normales

1. Créez une boucle for
2. Définissez une variable de comptage (i, dans ce cas) sur une matrice de valeurs 1-10. Cette variable de comptage sera utilisée pour appeler chaque image individuellement
3. Prenez l'élément i dans la matrice d'images pour extraire et convertir le nom de l'image d'une chaîne en un nombre à l'aide de la fonction num2str.
Trouvez le nombre de chiffres présents dans le nom de l'image à l'aide de la fonction numel. Affectez cette valeur à une variable, digits_normal. Ce nombre doit être 1 pour les nombres à un chiffre, 2 pour les nombres à deux chiffres et 3 pour les nombres à trois chiffres. Ces informations seront utilisées pour appeler automatiquement les images.
Étape 10: créer une boucle for pour télécharger automatiquement des images normales (suite)

3. Créez une instruction if contenant les trois possibilités des étapes précédentes. Si le nom de l'image a 1 chiffre, l'image sera appelée "im000", si elle a 2 chiffres, l'image sera appelée "im00", et si elle en a 3, l'image sera appelée "im0".
4. Sous chaque instruction if, attribuez une variable à imread « im » sous l'instruction if correspondante avec le nombre approprié de zéros (comme décrit ci-dessus), suivie du i.
Étape 11: recadrer les bordures de l'image
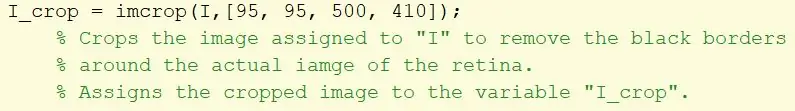
Prenez l'image d'origine et appliquez un filtre imcrop pour éliminer les bordures noires et assignez à une variable I_crop. Le rectangle de recadrage est spécifié à l'aide d'une matrice [95, 95, 500, 410].
Étape 12: Créer une image en niveaux de gris

Prenez l'image recadrée et appliquez le filtre rbg2gray pour changer l'image en niveaux de gris. Affectez cette image à la variable I2.
Étape 13: Créez une image contrastée
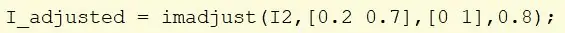
Prenez l'image I2 et utilisez imadjust pour redimensionner les valeurs d'intensité.
Prenez des valeurs comprises dans la plage [0.2, 0.7] et redimensionnez-les à [0, 1]. Le gamma est réglé sur 0,8 pour rendre l'image plus lumineuse. Affectez la nouvelle image à I_adjusted.
Étape 14: Améliorer l'image de contraste
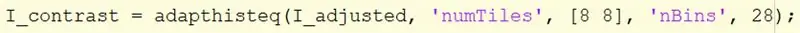
Prenez l'image I_adjusted et utilisez la fonction adapthisteq pour améliorer le contraste.
La syntaxe Adapthisteq requiert le nom de l'image, I_adjusted, « numTiles », la taille de numTiles, « nBins » et le nombre de bacs. La taille de numTiles est définie sur [8 8], divisant l'image en tuiles 8x8 et le nombre de cases est défini sur 28. Affectez l'image à I_constrast.
Étape 15: créer un filtre moyen

Créez une variable nommée 'meanfilt' en utilisant la fonction fspecial. Saisissez la « fonction moyenne » pour créer le filtre de moyenne et insérez [90 90] pour la taille de la fenêtre glissante.
Étape 16: Combinez le filtre de moyenne avec l'image contrastée
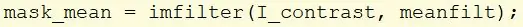
Créez une nouvelle variable nommée mask_mean et utilisez la fonction imfilter pour prendre l'image I_contrast et appliquer le filtre moyen créé précédemment.
Étape 17: Créer un nouveau masque moyen en soustrayant des pixels

Créez une variable nommée mask_mean2 et utilisez la fonction imsubtract pour soustraire la valeur de chaque pixel dans I_contrast du pixel correspondant dans mask_mean.
Étape 18: créer une image filtrée binaire

Transformez les images en niveaux de gris en noir et blanc en utilisant imbinarize. Entrée mask_mean2, 'adaptatif', 'ForegroundPolarity', 'dark', 'Sensitivity', 0.6. Attribuez cette nouvelle image à mask_binarize.
Étape 19: Supprimer les petits blobs trouvés dans les images filtrées
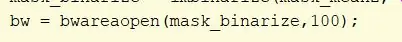
Supprimez les objets dont la connectivité est inférieure à 100 pixels à l'aide de la fonction bwareaopen sur mask_binarize et définissez la valeur seuil sur 100. Affectez la variable comme bw.
Étape 20: Créer un élément de structuration de disque

Créez un élément structurant de disque (avec un rayon de 2) à l'aide de la fonction strel. Attribuez-le à se.
Étape 21: Effectuer des opérations de fermeture morphologique

Prenez bw et appliquez la fonction imclose à l'élément structurel pour effectuer une opération de fermeture morphologique sur l'objet.
Étape 22: Trouvez les objets avec une connectivité d'au moins 8
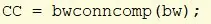
Prenez bw et utilisez bwconncomp pour trouver les objets avec une connectivité d'au moins 8 dans l'image. Attribuez le numéro de sortie à cc_1.
Étape 23: Trouvez le nombre maximum de pixels connectés
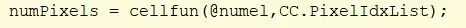
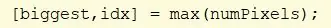
Utilisez la fonction cellfun pour exécuter la fonction "namel" sur chaque cellule de CC. Ceci trouve le nombre d'éléments dans la cellule PixelIdxList. Attribuez une valeur à « numPixels ».
Trouvez les valeurs maximales dans numPIxels. Attribuez le plus grand maximum à « plus grand » et l'indice de la valeur maximale à « idx ».
Étape 24: définissez les valeurs de pixels maximales sur 0 et recherchez les pixels avec une connectivité >=26 pixels
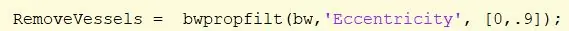



Réglez les pixels avec les plus grandes valeurs dans l'image "bw" à 0, rendant les pixels noirs.
Trouvez les objets avec une connectivité d'au moins 26 pixels dans l'image en utilisant bwconncomp. Affectez à la variable cc_1.
Étape 25: Supprimer les vaisseaux sanguins dans l'image
Retirez les vaisseaux sanguins encore présents dans l'image à l'aide de la fonction bwpropfilt avec une plage de [0, 0,9].
[0.9, 1] est exclu car les valeurs proches de 1 indiquent une ligne. Attribuer à « Supprimer les navires ».
Étape 26: Affichage des chiffres
Affichez chaque image filtrée dans une sous-intrigue. Imshow. avec les entrées « border » et « tight », affiche chaque image dans une structure de sous-parcelle. Ajoutez un titre à chaque image pour distinguer le filtre utilisé.
Étape 27: Retirez les vaisseaux et comptez les gouttes de sang
1. Prenez "RemoveVessels" et appliquez la fonction "Centroid" dans regionprops pour identifier les centroïdes des objets dans l'image. Ces objets doivent correspondre aux caillots sanguins présents dans l'image.
2. Comptez le nombre de caillots sanguins identifiés en prenant la longueur de la matrice centroïde.
Étape 28: Diagnostiquer l'image rétinienne en fonction du nombre de caillots sanguins identifiés
Utilisez les instructions if pour diagnostiquer l'image en fonction du nombre de caillots sanguins identifiés.
Si le nombre de centroïdes identifiés était inférieur ou égal à 5, l'image était identifiée comme normale.
Si le nombre de centroïdes était supérieur à 5, l'image était diagnostiquée avec une rétinopathie diabétique.
Le résultat est imprimé dans la fenêtre de commande à l'aide de fprintf.
Étape 29: S'il y a plus de 5 blobs…
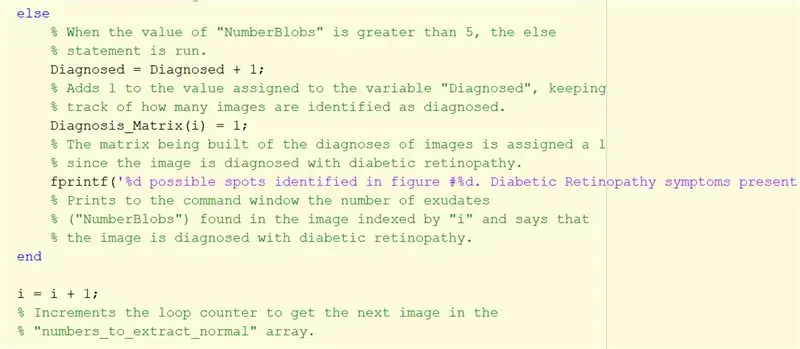
Répétez les instructions ci-dessus pour les images diagnostiquées en tant qu'instruction else. Cette partie s'exécutera si le nombre de blobs est supérieur à 5.
Terminez l'instruction if.
Étape 30: Répétez le processus de filtrage pour les images normales avec des valeurs de chiffre d'image comme 2 et 3
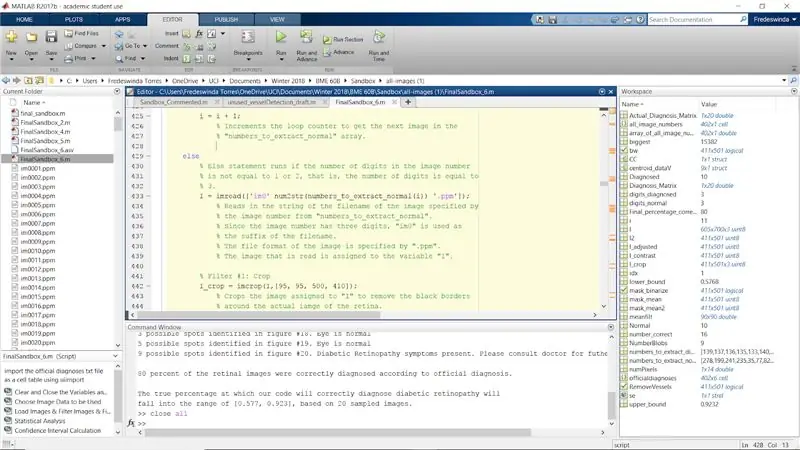
Répétez le processus pour le reste des instructions if d'origine lorsque numel (le nombre de chiffres dans le numéro d'image) est égal à 2 et 3. Ceci termine la boucle for pour les images normales.
Terminez la boucle for.
Étape 31: Répétez le processus entier pour les images diagnostiquées
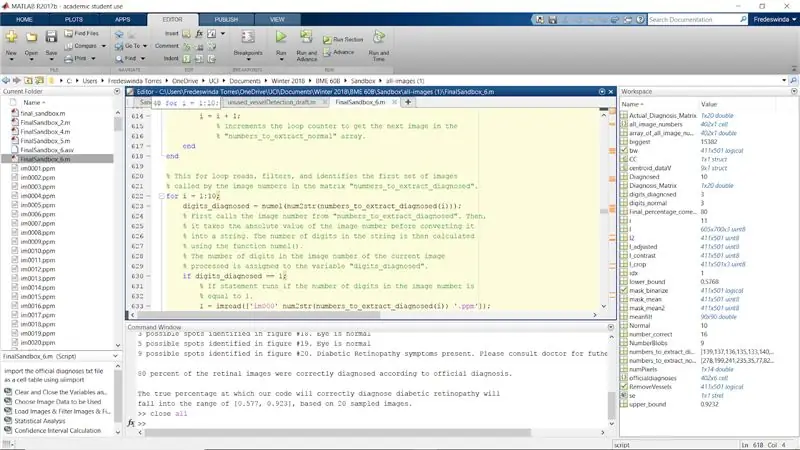
Répétez l'ensemble du processus en utilisant les images diagnostiquées répertoriées par la matrice « numbers_to_extract_diagnosed ».
Assurez-vous de parcourir chaque chiffre (i) et de le changer en chiffre (i + 10) afin que les chiffres diagnostiqués apparaissent sous forme d'images 11 à 20.
Étape 32: Analyse statistique
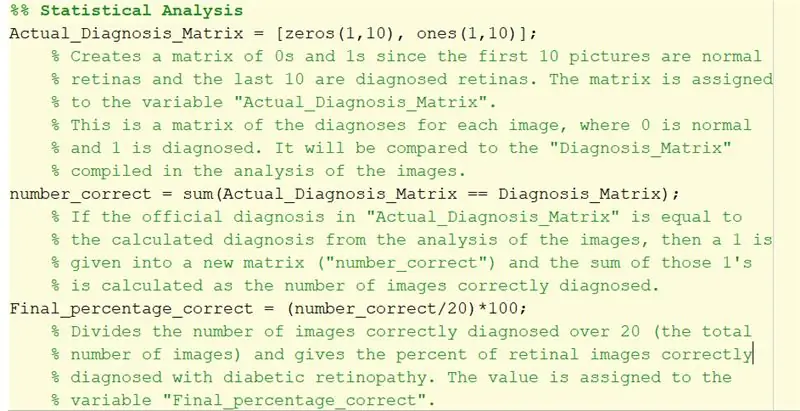
1. 'Actual_Diagnosis_Matrix' est utilisé pour comparer les résultats au diagnostic officiel trouvé dans le fichier txt. Les 10 premiers zéros indiquent que les 10 premières images doivent être normales. Les 10 dernières indiquent que les 10 dernières images doivent être classées comme rétinopathie diabétique.
2. Le double signe égal utilisé pour créer 'number_correct' crée un tableau logique en comparant la valeur des éléments correspondants de 'Actual_Diagnosis_Matrix' à 'Diagnosis_Matrix' créé à partir de la boucle for.
Pour chaque élément qui correspond au diagnostic, un 1 sera ajouté, ce qui signifie que le code a correctement diagnostiqué cette image. S'il est incorrect, il ajoutera un 0 à la matrice.
Ensuite, en faisant la somme de cela, on additionne tous les uns. En d'autres termes, il trouve la somme des images correctement diagnostiquées.
3. 'Final_percentage_correct' est le pourcentage calculé de la précision du code diagnostiqué pour la rétinopathie diabétique. Le nombre d'images diagnostiquées correctement est divisé par 20 (le nombre total d'images) et multiplié par 100 pour trouver le pourcentage de diagnostics réussis.
Étape 33: Trouver l'intervalle de confiance
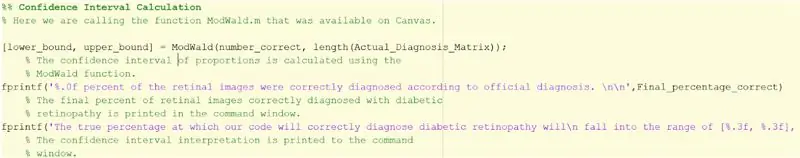
1. Assurez-vous d'avoir téléchargé ModWald.m pour l'appeler en tant que fonction. Sans la fonction, vous devriez calculer vous-même l'intervalle de confiance en utilisant la méthode de Wald modifiée.
2. La fonction ModWald a 2 entrées où la première est le nombre d'images correctement identifiées, et la seconde est le nombre total d'images.
3. La fonction ModWald affichera les limites inférieure et supérieure de l'intervalle de confiance des proportions pour la précision des données échantillonnées. En d'autres termes, cela vous donnera un intervalle de pourcentages où se situera le vrai pourcentage de l'exactitude du code.
4. Utilisez fprintf ci-dessous pour afficher les statistiques et l'intervalle de confiance dans la fenêtre de commande.
> fprintf('%.0f % des images rétiniennes ont été correctement diagnostiquées selon le diagnostic officiel. \n\n', Final_percentage_correct)
> fprintf('Le pourcentage réel auquel notre code diagnostiquera correctement la rétinopathie diabétique se situera\n dans la plage de [%.3f, %.3f], sur la base de 20 images échantillonnées \n', lower_bound, upper_bound)
Conseillé:
Crédit supplémentaire de projet final ECG-BME 305 automatisé : 7 étapes

ECG automatisé - BME 305 Projet final Crédit supplémentaire : Un électrocardiogramme (ECG ou ECG) est utilisé pour mesurer les signaux électriques produits par un cœur qui bat et il joue un rôle important dans le diagnostic et le pronostic des maladies cardiovasculaires. Certaines des informations obtenues à partir d'un ECG incluent le rythme
Modèle de circuit ECG automatisé : 4 étapes
Modèle de circuit ECG automatisé : l'objectif de ce projet est de créer un modèle de circuit avec plusieurs composants qui peuvent amplifier et filtrer de manière adéquate un signal ECG entrant. Trois composants seront modélisés individuellement : un amplificateur d'instrumentation, un filtre coupe-bande actif, et un
Aide au stationnement Réparation/diagnostic facile : 4 étapes

Aide au stationnement Réparation/Diagnostic faciles : Ok, commençons, j'ai une Chevrolet Avalanche 2010 et elle a 4 capteurs d'aide au stationnement dans son pare-chocs arrière. Cet intraitable peut être utilisé sur et sur un véhicule au meilleur de ma connaissance, que vous ayez l'avant ou Rea ou les deux. Alors je suis allé chez mon préféré
Disques durs : diagnostic, dépannage et maintenance : 3 étapes

Disques durs : diagnostic, dépannage et maintenance : qu'est-ce qu'un disque dur ? - En termes simples, le disque dur est ce qui stocke toutes vos données. Il abrite le disque dur, où se trouvent physiquement tous vos fichiers et dossiers. Les informations sont stockées magnétiquement sur le disque, de sorte qu'elles restent sur le lecteur même lorsque
Guide de codage/diagnostic BMW INPA E60 : 4 étapes

Guide de codage / diagnostic BMW INPA E60: Dans cet article, je vais collecter quelques cas concernant le cas de travail BMW INPA sur BMW E60, et si vous avez d'autres cas BMW INPA E60 que vous souhaitez partager avec les gens, veuillez les envoyer à mon e-mail. je le mettrai ici pour le partager
